Онкогеномика
Онкогеномика ― потполе на геномиката што ги карактеризира гените поврзани со ракот. Насочена е на геномски, епигеномски и транскриптни промени кај ракот.
Ракот е генетско заболување предизвикано од акумулација на мутации на ДНК и епигенетски промени што доведуваат до неограничена клеточна пролиферација и создавање неоплазми. Целта на онкогеномиката е да бидат идентификувани нови онкогени или туморски потиснувачки гени кои можат да дадат нови сознанија за дијагнозата на ракот, предвидувајќи го клиничкиот исход на ракот и нови цели за терапии за рак. Успехот на целните терапии за рак, како што се иматинивот, трастузумабот и бевацизумабот, ја зголемил надежта за онкогеномиката да разјасни нови цели за лекување на ракот.[1]

Покрај разбирањето на основните генетски механизми кои започнуваат или поттикнуваат напредок на ракот, онкогеномиката цели на личен третман за рак. Ракот се развива поради мутации на ДНК и епигенетски промени кои се собирани случајно. Идентификувањето и насочувањето на мутациите кај поединечен пациент може да доведе до зголемена ефикасност на третманот.
Завршувањето на Проектот за човечкиот геном го олеснило полето на онкогеномиката и ги зголемило способностите на истражувачите да пронајдат онкогени. Технологиите за секвенционирање и општите техники за профилирање на метилација се применувани во проучувањето на онкогеномиката.
Историја[уреди | уреди извор]
Времето на геномиката започнало во 1990-тите, со создавање на секвенци на ДНК на многу организми. Во 21 век, завршувањето на Проектот за човечкиот геном овозможило проучување на функционалната геномика и испитување на туморски геноми. Ракот е главен фокус.
Времето на епигеномиката во голема мера започнала неодамна, околу 2000 година.[2][3] Еден главен извор на епигенетски промени е изменетата метилација на цитозинско-гванинските острови кај промоторскиот регион на гените (види метилација на ДНК кај рак). Голем број на неодамна осмислени методи можат да го проценат статусот на метилација на ДНК кај ракот наспроти нормалните ткива.[4] Некои методи ја проценуваат метилацијата на цитозини-гванини сместени во различни класи на локуси, вклучително и цитозинско-гванински острови, брегови, како и промотори, генски тела и меѓугенски региони.[5] Ракот е исто така главен фокус на епигенетските студии.
Пристапот до секвенционирање на целиот геном на ракот е важен за истражувањето на ракот (или геномот на ракот), бидејќи:
- Мутациите се непосредна причина за рак и го дефинираат фенотипот на туморот.
- Пристапот до примероци од ракородни и нормални ткива од ист пациент и фактот дека повеќето мутации на рак претставуваат соматски настани, овозможуваат идентификација на мутации специфични за ракот.
- Мутациите на ракот се собирани и понекогаш се поврзани со фазата на болеста. Се разликувани метастази и отпорност на лекови.[6]
Пристапот до профилирање на метилација е важен за истражување на ракот бидејќи:
- Епигенетскиот ген, заедно со мутацискиот ген, можат да дејствуваат како непосредни причинители на рак[7]
- Епимутациите на ракот се собирани и понекогаш се поврзани со фазата на болеста[8]
Секвенционирање на целиот геном[уреди | уреди извор]
Првиот геном со рак бил секвенциониран во 2008 година.[6] Оваа студија секвенционира вообичаен геном на акутна миелоидна леукемија и неговиот нормален геном добиен од истиот пациент. Споредбата открила десет мутирани гени. Веќе било сметано дека два придонесуваат за напредокот на туморот: внатрешно тандемско удвојување на генот на тирозинска киназа на рецепторот FLT3, кој го активира сигнализирањето на киназата и е поврзан со лоша прогноза и вметнување четири бази во егзонот 12 на генот NPM1 (нуклеофозмин ц). Овие мутации се наоѓаат во 25-30% од туморите на акутната миелоидна леукемија и е сметано дека придонесуваат за напредокот на болеста наместо директно да ја предизвикаат.
Останатите 8 биле нови мутации и сите биле промени во една база: четири биле во семејства кои се силно поврзани со патогенезата на ракот (PTPRT, CDH24, PCLKC и SLC15A1). Останатите четири немале претходна поврзаност со патогенезата на ракот. Тие навистина имале потенцијални функции во метаболичките патишта кои наведувале механизми со кои тие би можеле да дејствуваат за да го промовираат ракот (KNDC1, GPR124, EB12, GRINC1B).
Овие гени се вклучени во патишта за кои е познато дека придонесуваат за патогенезата на ракот, но пред оваа студија повеќето не би биле кандидати за насочена генска терапија. Оваа анализа го потврдила пристапот на секвенционирање на геномот на целиот рак во идентификувањето на соматските мутации и важноста на напореднито секвенционирање на геномите на нормалните и туморските клетки.[9]
Во 2011 година, геномот на исклучителен пациент со рак на мочниот меур чиј тумор бил елиминиран со лекот еверолимус бил секвенциониран, откривајќи мутации во два гени, TSC1 и NF2. Мутациите ја дерегулирале mTOR, белковината инхибиран од еверолимус, овозможувајќи му да се размножува без ограничување. Како резултат на тоа, во 2015 година, во Националниот институт за рак на Соединетите Држави, била создадена Иницијативата за исклучителни одговори. Иницијативата им овозможува на таквите исклучителни пациенти (кои позитивно реагирале најмалку шест месеци на лек за рак кој обично не успева) да ги секвенционираат нивните геноми за да бидат идентификувани релевантните мутации. Откако ќе бидат идентификувани, другите пациенти би можеле да бидат скринирани за тие мутации и потоа да им се даде лекот. Во 2016 година За таа цел, во 2015 година започнало национално испитување на лекови за рак, во кое биле вклучени до дваесет и четиристотини центри. Пациентите со соодветни мутации се совпаѓани со еден од повеќе од четириесет лекови.[10]
Во 2014 година, Центарот за молекуларна онкологија го промовирал тестот MSK-IMPACT, алатка за преглед што бара мутации во 341 ген поврзани со ракот. До 2015 година биле прегледани повеќе од пет илјади пациенти. Пациентите со соодветни мутации имаат право да се запишат во клинички испитувања кои обезбедуваат насочена терапија.[10]
Технологии[уреди | уреди извор]
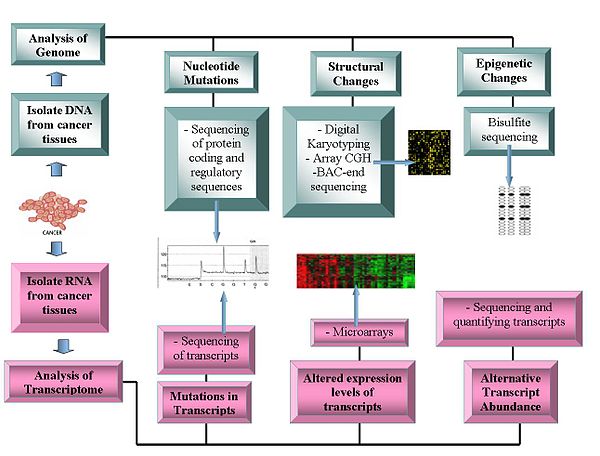
Геномичките технологии се:
Секвенционирање на геномот[уреди | уреди извор]
- Секвенционирање на ДНК: Секвенционерите засновани на пиросеквенционирање нудат релативно евтин метод за создавање податоци за секвенцата.[1][11][12]
- Споредбена геномска хибридизација на низа: Оваа техника ги мери разликите во бројот на копии на ДНК помеѓу нормалниот и ракородниот геном. Го користи интензитетот на флуоресценција од примероците означени со флуоресцентни, кои се хибридизираат со познати сонди на микрочип.[13][14]
- Анализа на претставителен олигонуклеотиден микрочип: Открива варијација на бројот на копии со помош на засилени геномски фрагменти сварени со ограничување кои се хибридизирани со човечки олигонуклеотиди, постигнувајќи резолуција помеѓу 30 и 35 kbit/с.[15]
- Дигитално кариотипирање: Открива варијација на бројот на копии со помош на геномични ознаки добиени преку варење со рестрикциони ензими. Овие ознаки потоа се поврзувани со дигитални ознаки, се спојувани, клонирани, секвенционирани и картирани назад во референтниот геном за да биде оценета густината на ознаката.[16][17]
- Завршно секвенционирање на бактериски вештачки хромозом (профилирање на завршна секвенца): Ги идентификува хромозомските точки на прекин со создавање на библиотека на бактериски вештачки хромозом од геном на рак и секвенционирање на нивните краеви. Клоновите со бактериски вештачки хромозом, кои содржат хромозомски аберации имаат завршни секвенци кои не се картирани на сличен регион на референтниот геном, со што е идентификувана хромозомската точка на прекин.[18]
Транскриптоми[уреди | уреди извор]
- Микрочипови: Проценка на изобилството на препис (транскриптом). Корисно во класификацијата, прогнозата, ја зголемува можноста за пристапи на диференцијален третман и помагаат во идентификација на мутации во регионите за кодирање на белковините.[19][20] Релативното изобилство на алтернативни преписи станала важна особина на истражувањето на ракот. Посебни алтернативни облици на транскрипт корелираат со специфични видови рак.[21]
- Секвенционирање на РНК
Биоинформатика и функционална анализа на онкогени[уреди | уреди извор]
Биоинформатичките технологии овозможуваат статистичка анализа на геномските податоци. Функционалните особини на онкогените допрва треба да бидат утврдени. Потенцијалните функции ги вклучуваат нивните преобразувачки способности кои се однесуваат на создавање тумор и специфични улоги во секоја фаза од развојот на ракот.
По откривањето на соматски мутации на рак во група примероци од рак, може да бидат спроведени биоинформатички сметачки анализи за да бидат идентификувани веројатните функционални и веројатни мутации на двигател. Постојат три главни пристапи кои рутински се користени за оваа идентификација: картирање на мутации, проценка на ефектот на мутација на функцијата на протеин или регулаторен елемент и пронаоѓање знаци на позитивно одбирање кај група тумори. Пристапите не се нужно последователни, меѓутоа, постојат важни односи на првенство помеѓу елементите од различните пристапи. На секој чекор се користени различни алатки.[22]
Оперомика[уреди | уреди извор]
Оперомиката има за цел да интегрира геномика, транскриптомика и протеомика за да ги разбере молекуларните механизми кои се во основата на развојот на ракот.[23]
Споредбена онкогеномика[уреди | уреди извор]
Споредбената онкогеномика користи споредби меѓу видовите за да бидат идентификувани онкогените. Ова истражување вклучува проучување на геномите на ракот, транскриптомите и протеомите кај моделните организми како што се глувците, идентификување на потенцијалните онкогени и упатување назад кон примероците на човечки рак за да биде видено дали хомолозите на овие онкогени се важни за предизвикување рак кај луѓето.[24] Генетските промени кај моделите на глувци се слични на оние кои се наоѓаат кај човечките ракови. Овие модели се создавани со методи, вклучувајќи мутагенеза на ретровирусно вметнување или пресадување на ракородни клетки.
Извор на мутации на двигател на ракот, мутагенеза на ракот[уреди | уреди извор]
Мутациите обезбедуваат суровина за природно одбирање во еволуцијата и можат да бидат предизвикани од грешки во репликацијата на ДНК, дејството на егзогените мутагени или ендогеното оштетување на ДНК. Машинеријата за репликација и одржување на геномот може да биде оштетена од мутации или изменета од физиолошки услови и диференцијални нивоа на изразување кај ракот.[25]
Како што е наведено од Гао и колегите,[26] стабилноста и интегритетот на човечкиот геном се одржувани со системот за одговор на оштетување на ДНК. Непоправеното оштетување на ДНК е главна причина за мутации кои предизвикуваат создавање рак (канцерогенеза).[27][28] Ако поправката на ДНК е дефицитарна, оштетувањето на ДНК има тежнеење да се насобира. Ваквото прекумерно оштетување на ДНК може да ги зголеми мутационите грешки за време на репликацијата на ДНК поради синтезата на транслезија склона кон грешки. Вишокот на оштетување на ДНК, исто така, може да ги зголеми епигенетските промени поради грешки при поправка на ДНК.[29][30] Ваквите мутации и епигенетски промени може да доведат до рак. Гените со одговор на оштетување на ДНК често се потиснати кај човечкиот рак со епигенетски механизми. Таквата репресија може да вклучи метилација на ДНК на промоторните региони или репресија на гените со одговор на оштетување на ДНК од микро РНК. Епигенетската репресија на гените со одговор на оштетување на ДНК се јавува почесто од генската мутација кај многу видови рак (види епигенетика на ракот). Така, епигенетската репресија често игра поважна улога од мутацијата во намалувањето на изразувањето на гените со одговор на оштетување на ДНК. Оваа намалена експресија на гените со одговор на оштетување на ДНК е веројатно важен двигател на создавањето рак.
Контекстот на нуклеотидната секвенца влијае на веројатноста за мутација[31][32][33] и анализата на мутационите (способни да мутираат) мотиви на ДНК може да биде од суштинско значење за разбирање на механизмите на мутагенеза кај ракот. Ваквите мотиви ги претставуваат отпечатоците од прстите на меѓудејствијата помеѓу ДНК и мутагените, помеѓу ДНК и ензимите за поправка/репликација/изменување. Примери на мотиви се мотивот со цитидин деаминаза индуцирана од активација WRCY/RGYW (W = A или T, R = пурин и Y = пиримидин) со C до T/G/A мутации,[33] и склони кон грешки на полимераза на ДНК на η атрибуирани мутации поврзани со цитидин деаминаза индуцирана од активација (A до G/C/G) во мотиви WA/TW.[34]
Друг (агностички) начин да бидат анализирани набљудуваните мутациски спектри и контекстот на секвенцата на ДНК на мутации во туморите вклучува здружување на сите мутации од различни врсти и контексти од примероците од рак во дискретна распространетост. Доколку се достапни повеќе примероци од рак, нивните мутации зависни од контекст може да бидат претставени во облик на ненегативна матрица. Оваа матрица може дополнително да се разложи на компоненти (мутациски потписи) кои идеално треба да ги опишат поединечните мутагени фактори.[35] Предложени се неколку сметачки методи за решавање на овој проблем на распаѓање. Првата примена на методот за факторизирање на не-негативна матрица е достапна во рамката за мутациски потпис на Сенгеровиот институт во облик на пакет на софтверот MATLAB (матрична лабораторија).[36] Од друга страна, ако мутациите од еден примерок на тумор се достапни само, пакетот DeconstructSigs R[37] и осплужувачот MutaGene[38] може да обезбедат идентификација на придонесите на различни мутациски потписи за еден примерок од тумор. Дополнително, опслужувачот MutaGene обезбедува мутагенски или специфични за рак модели и потписи на мутациона позадина што може да бидат применети за да биде пресметана очекуваната мутабилност на ДНК и белковинското место за да бидат раздвоени релативните придонеси на мутагенезата и одбирање во создавањето рак.
Синтетичка смртност[уреди | уреди извор]
Синтетичката смртност настанува кога комбинацијата на недостатоци во изразувањето на два или повеќе гени доведува до клеточна смрт, додека недостаток на само еден од овие гени не. Недостатоците може да настанат преку мутации, епигенетски промени или инхибитори на еден од гените.
Терапевтскиот потенцијал на синтетичката смртност како ефикасна противракородна стратегија постојано е подобрувана. Неодамна, применливоста на синтетичката смртност за насочена терапија против рак била зголемена поради неодамнешната работа на научниците, вклучително и Роналд А. Депињо и колегите, во она што се нарекува „колатерална смртност“. Мулер и колегите откриле дека патничките гени, со хромозомска близина до туморските потиснувачки гени, се колатерално избришани кај некои видови рак.[39] Така, идентификацијата на колатерално избришаните непотребни гени кои вршат суштинска клеточна функција може да биде неискористениот резервоар за потоа да биде следен пристапот на синтетичка смртност. Затоа, колатералната смртност има голем потенцијал во идентификацијата на нови и одбрани терапевтски цели во онкологијата.[40] Во 2012 година, Мулер и колегите идентификувале дека хомозиготното бришење на вишокот есенцијален гликолитички ген ENO1 кај човечкиот глиобластом е последица на близината до бришењата на локусот на туморските потиснувачи 1p36 и може да има потенцијал за пристап на синтетичка смртност кон инхибиција на глиобластомот.[39] ENO1 е еден од трите хомологни гени (ENO2, ENO3) кој го кодира ензимот алфа-енолаза кај цицачите.[41] ENO2, која ја кодира енолазата 2, најмногу е изразуван во нервните ткива, што доведува до постулација дека во глиобластомите избришани од ENO1, ENO2 може да биде идеална цел како вишок хомолог на ENO1.[42] Мулер открил дека и генетската и фармаколошката инхибиција на ENO2 во глуобластомските клетки со хомозиготно бришење на ENO1 предизвикува синтетички исход на смртност со селективно убивање на глиобластомските клетки.[39] Во 2016 година, Мулер и неговите колеги го откриле антибиотикот SF2312 како инхибитор на енолаза со високо наномоларен опсег кој преференцијално ја инхибира пролиферацијата на клетките на глиомот и гликолитичкиот флукс во клетките избришани од ENO1.[43] Било покажано дека SF2312 е поефикасен од инхибиторот на пан-енолаза PhAH и има поголема специфичност за инхибиција на ENO2 во однос на ENO1.[43] Последователната работа на истата работна група, покажала дека истиот пристап може да биде применет и за ракот на панкреасот, при што хомозиготно избришаниот SMAD4 резултира со колатерално бришење на митохондрискиот малатен ензим 2 (ME2), оксидативна декарбоксилаза есенцијална за редукционата хомеостаза.[44] Деј и колегите покажувале дека геномското бришење на МЕ2 во клетките на аденокарцином на дукталниот панкреас резултира со високи ендогени реактивни кислородни видови, доследни со ракот на панкреасот поттикнат од KRAS, и суштински ги подготвува ME2-нултаните клетки за синтетичка смртност со намалување на вишокот NAD(P)+-зависна изоформа. Утврдено е дека ефектите од намалувањето на ME3 се посредувани со инхибиција на „de novo“ синтезата на нуклеотидите кои произлегуваат од активирањето на AMPK и апоптозата посредувана од митохондриските реактивни кислородни видови.[45][44] Во меѓувреме, Ојке и колегите ја покажале генерализираноста на концептот со целење на вишок есенцијални гени во постапка различна од метаболизмот, имено SMARCA4 и SMARCA2 подединици во комплексот SWI/SNF за ремоделирање на хроматин.[46]
Некои онкогени се од суштинско значење за опстанокот на сите клетки (не само клетките на ракот). Така, лековите кои ги исфрлаат овие онкогени (а со тоа ги убиваат клетките на ракот), исто така, може да ги оштетат нормалните клетки, предизвикувајќи значителна болест. Сепак, други гени може да бидат од суштинско значење за клетките на ракот, но не и за здравите клетки.
Третманите засновани на начелото на синтетичка смртност го продолжиле преживувањето на пациентите со рак и ветуваат за иден напредок во промената на канцерогенезата. Главниот вид синтетичка смртност работи на дефектот за поправка на ДНК што често започнува рак и сè уште е присутен во клетките на туморот. Некои примери се дадени овде.
Изразувањето на BRCA1 или BRCA2 е во недостаток кај повеќето висококвалитетни ракови на дојка и јајници, обично поради епигенетска метилација на неговиот промотор или епигенетска репресија од прекумерно изразена микро РНК (видете во статиите BRCA1 и BRCA2). BRCA1 и BRCA2 се важни компоненти на главната патека за хомологно рекомбинирачко поправка на прекините со двојни соеви. Ако едните или другите се во недостаток, тоа го зголемува ризикот од рак, особено рак на дојка или рак на јајниците. Резервниот пат за поправка на ДНК, за некои од оштетувањата што обично се поправани со BRCA1 и BRCA2, зависи од PARP1. Така, многу видови на рак на јајниците реагираат на третман одобрен од Службата за храна и лекови на Соединетите Држави, со PARP инхибитор, предизвикувајќи синтетичка смртност на клетките на ракот со недостаток на BRCA1 или BRCA2. Овој третман, исто така, е оценуван за рак на дојка и бројни други видови рак во клиничките испитувања во фаза III во 2016 година.[47]
Постојат два патишта за хомологно рекомбинациско поправање на прекините со двојни соеви. Главниот пат зависи од BRCA1, PALB2 и BRCA2 додека алтернативната патека зависи од RAD52.[48] Предклиничките студии, кои вклучуваат епигенетски намалени или мутирани клетки со недостаток на BRCA (во култура или вбризгувани во глувци), покажуваат дека инхибицијата на RAD52 е синтетички смртоносна со недостаток на BRCA.[49]
Мутациите во гените кои се користени за поправка на несовпадната ДНК предизвикуваат висока стапка на мутација.[50] Кај туморите, таквите чести последователни мутации често создаваат „не-само“ имуногени антигени. Клиничко испитување во Фаза II со луѓе, со 41 пациент, евалуирало еден синтетички смртоносен пристап за тумори со или без дефекти на поправка на несовпадната ДНК.[51] Производот на генот PD-1 вообичаено ги потиснува цитотоксичните имунолошки одговори. Инхибицијата на овој ген овозможува поголем имунолошки одговор. Кога пациентите со рак со дефект во поправка на несовпадната ДНК во нивните тумори биле изложени на инхибитор на PD-1, 67-78% од пациентите доживеале преживување без напредок поврзано со имунолошкиот систем. Спротивно на тоа, за пациентите без дефектна поправка на несовпадната ДНК, додавањето на PD-1 инхибитор создало само 11% од пациентите со имуно поврзано преживување без напредок. Така, инхибицијата на PD-1 е примарно синтетички смртоносна со дефекти на поправка на несовпадната ДНК.
ARID1A, модификатор на хроматин, е потребен за нехомологно поврзување на краевите, главна патека која ги поправа прекините на двојните соеви во ДНК,[52] и исто така има регулаторни улоги на транскрипцијата.[53] ARID1A мутациите се една од 12-те најчести ракородни мутации.[54] Мутација или епигенетско намалено изразување[55] на ARID1A е пронајдено кај 17 врсти рак.[56] Предклиничките студии кај клетки и кај глувци покажуваат дека синтетичката смртност за недостаток на ARID1A се јавува или со инхибиција на активноста на метилтрансферазата на EZH2,[57][58] или со додавање на киназен инхибиторски дасатиниб.[59]
Друг пристап е поединечно да биде исфрлен секој ген во геномот и да биде набљудуван ефектот врз нормалните и ракородните клетки.[60][61] Ако соборувањето на инаку несуштински ген има мало или никакво влијание врз здравите клетки, но е смртоносно за ракородните клетки кои содржат мутиран онкоген, тогаш сузбивањето на потиснатиот ген низ целиот систем може да ги уништи ракородните клетки, додека здравите да останат релативно неоштетени. Техниката била искористена за да бидат идентификувани инхибиторите на PARP-1 за лекување на ракови поврзани со BRCA1/BRCA2.[62][63] Во овој случај, комбинираното присуство на PARP-1 инхибиција и на мутациите поврзани со ракот во гените со BRCA е смртоносно само за ракородните клетки.
Бази на податоци за истражување на ракот[уреди | уреди извор]
Проектот за геномот на ракот е иницијатива за картирање на сите соматски мутации кај ракот. Проектот систематски ги секвенцира егзоните и страничните споеви на геномите на примарните тумори и ракородните клеточни линии. Софтверот COSMIC ги прикажува податоците создадени од овие опити. Од февруари 2008 година, Проектот идентификувал 4.746 гени и 2.985 мутации во 1.848 тумори.
Проектот за анатомијата на геномот на ракот вклучува информации за истражување на геномите, транскриптомите и протеомите на ракот.
Progenetix е онкогеномска референтна база на податоци, која ги претставува цитогенетските и молекуларно-цитогенетските податоци за туморот.
Oncomine собрал податоци од профилите на транскриптом на ракот.
Интегративната база на податоци за онкогеномика IntOGen и сетови на податоци Gitools интегрираат повеќедимензионални човечки онкогеномски податоци класифицирани по вид тумор. Првата верзија на IntOGen била насочена на улогата на дерегулирана генско изразување и варијацијата на бројот на копии кај ракот.[64] Подоцнежната верзија ги нагласила мутационите гени за рак на 28 видови тумори.[65][66] Сите изданија на податоци за IntOGen се достапни во нивната база на податоци.
Меѓународниот конзорциум за геном на рак е најголемиот проект за собирање податоци за човечкиот геном на рак. До податоците се достапни преку мрежната страница на Конзорциумот. BioExpress® Oncology Suite содржи податоци за генско изразување од примарни, метастатски и бенигни примероци на тумори и нормални примероци, вклучително и соодветните соседни контроли. Пакетот вклучува примероци од хематолошки малигнитет за многу познати видови рак.
Специфичните бази на податоци за моделни животни ја вклучуваат Базата на податоци за гените за рак со ознака за ретровируси која составила истражување за ретровирусна и вметната транспозонска мутагенеза кај тумори на глувци.
Генски семејства[уреди | уреди извор]
Мутациона анализа на цели генски семејства открила дека гените од исто семејство имаат слични функции, како што е предвидено со слични кодирани секвенци и белковински домени. Две такви класи се семејството кинази, вклучено во додавање фосфатни групи на белковините и фосфатазното семејство, вклучено во отстранувањето на фосфатните групи од белковините.[67] Овие семејства прво биле испитани поради нивната очигледна улога во трансдукцијата на клеточните сигнали за клеточен раст или смрт. Особено, повеќе од 50% од раковите на дебелото црево носат мутација во генот на киназа или фосфатаза. Генот на фосфатидилинозитолд 3-кинази (PIK3CA) ги кодира липидните кинази кои вообичаено содржат мутации во ракот на дебелото црево, дојката, желудникот, белите дробови и разни други ракови.[68][69] Терапиите со лекови може да го инхибираат PIK3CA. Друг пример е генот BRAF, еден од првите кои се вмешани во меланоми.[70] BRAF шифрира серинско/треонинска киназа која е вклучена во сигналниот пат за раст на RAS-RAF-MAPK. Мутациите во BRAF предизвикуваат конститутивна фосфорилација и активност кај 59% од меланомите. Пред BRAF, генетскиот механизам на развој на меланомот бил непознат и затоа прогнозата за пациентите била лоша.[71]
Митохондриска ДНК[уреди | уреди извор]
Мутациите на митохондриската ДНК се поврзани со образување тумори. Идентификувани биле четири вида мутации на митохондриската ДНК:[72]
Точкести мутации[уреди | уреди извор]
Точкести мутации биле забележани во кодирањето и некодирачкиот регион на митохондриската ДНК содржана во клетките на ракот. Кај поединци со рак на мочниот меур, главата/вратот и белите дробови, точките мутации во областа на кодирање покажуваат знаци на слични едни на други. Ова наведува дека кога здравата клетка се преобразува во туморска клетка (неопластична преобразба) изгледа дека митохондриите стануваат хомогени. Обилните точкестии мутации сместени во некодирачкиот регион, Д-јамка, на ракородните митохондрии наведуваат дека мутациите во овој регион може да бидат важна особина кај некои видови рак.[72]
Бришења[уреди | уреди извор]
Овој вид мутација спорадично е откриван поради неговата мала големина (< 1kb). Појавата на одредени специфични мутации на митохондриската ДНК (бришење од 264 bp и бришење од 66 bp во комплексот 1 на подединицата на генот ND1) кај повеќе видови рак обезбедува одредени докази дека малите бришења на митохондриската ДНК може да бидат појавени на почетокот на создавањето на туморот (туморигенеза). Исто така, наведува дека количината на митохондриите што ги содржат овие бришења се зголемувани како што туморот напредува. Исклучок е релативно големото бришење кое се појавува кај многу видови рак (познато како „заедничко бришење“), но повеќе бришења на митохондриска ДНК во големи размери се пронајдени во нормалните клетки во споредба со туморските клеткит. Ова може да се должи на навидум приспособната постапка на клетките на туморот за елиминирање на сите митохондрии кои ги содржат овие големи бришења („вообичаеното бришење“ е > 4kb).[72]
Вметнувања[уреди | уреди извор]
Две мали вметнувања на митохондриската ДНК од ~260 и ~520 bp може да бидат присутни кај рак на дојка, рак на желудник, рак на црниот дроб и рак на дебелото црево и во нормални клетки. Не е воспоставена корелација помеѓу овие вметнувања и ракот.[73]
Мутации на бројот на копии[уреди | уреди извор]
Карактеризацијата на митохондриската ДНК преку анализи на верижна реакција на полимераза во вистинско време покажува присуство на квантитативна промена на бројот на копии на митохондриската ДНК кај многу видови рак. Очекувано е зголемување на бројот на копии поради оксидативниот стрес. Од друга страна, намалувањето се смета дека е предизвикано од соматски точкести мутации во местото на потекло на репликацијата на тешкиот сој и/или D310 хомополимерниот ц-истегнување во регионот на Д-јамката, мутации во p53 (туморски сузбивачки ген) посредуван пат и/или неефикасна ензимска активност поради мутации на полимеразна подединица гама на ДНК. Секое зголемување/намалување на бројот на копии тогаш останува постојан во клетките на туморот. Фактот дека количината на митохондриската ДНК е постојана во клетките на туморот наведува дека количината на митохондриската ДНК е контролирана од многу посложен систем во клетките на туморот, наместо едноставно да биде менуван како последица на абнормална клеточна пролиферација. Улогата на содржината на митохондриската ДНК кај човечките ракови очигледно варира за одредени видови или места со тумори.[72]
| Вид рак | Место на точкасти мутации | Нуклеотидна положба на бришења | Зголемување на копијата на митохондриската ДНК # | Намалување на копијата на митохондриската ДНК # | |||
|---|---|---|---|---|---|---|---|
| Д-јамка | Информациска РНК | Преносна РНК | Рибозомна РНК | ||||
| Мочен меур[74] | X | X | X | 15.642-15.662 | |||
| Гради[75][76][77][78] | X | X | X | X | 8470-13,447 и 8482-13459 | X | |
| Глава и врат[75][79][80] | X | X | X | X | 8470-13,447 и 8482-13459 | X | |
| Уста[81] | X | X | 8470-13,447 и 8482-13459 | ||||
| Црн дроб[82][83] | X | X | X | X | 306-556 и 3894-3960 | X | |
| Хранопровод[84] | X | X | X | 8470-13,447 и 8482-13459 | X | ||
| Желудник[85] [86][87] | X | X | X | 298-348 | X | ||
| Простата[88][89] | X | X | 8470-13,447 и 8482-13459 | X | |||
57,7% (500/867) содржеле безбројни соматски точки и од 1172 испитувани мутации 37,8% (443/1127) биле најдени во контролниот регион на Д-јамката, 13,1% (154/1172) биле најдени во гените на преносната РНК или рибозомната РНК и 49,1% (575/1127) се пронајдени во гените на информациската РНК потребни за производство на комплекси потребни за митохондриско дишење.
Дијагностички примени[уреди | уреди извор]
Некои противракородни лекови се насочени кон митохондриската ДНК и покажале позитивни резултати во убивањето на клетките на туморот. Истражувањата користеле митохондриски мутации како биомаркери за терапија со ракородни клетки. Полесно е да биде целена мутацијата во митохондриската ДНК наспроти јадрената ДНК бидејќи митохондрискиот геном е многу помал и полесен за проверка за специфични мутации. Промените на содржината на митохондриската ДНК пронајдени во примероците крв може да послужат како прегледен маркер за предвидување на идната подложност на рак, како и следење на напредокот на малигниот тумор. Заедно со овие потенцијални корисни особини на митохондриската ДНК, таа не е под контрола на клеточниот циклус и е важен за одржување на создавањето на аденозин трифостат и митохондриската хомеостаза. Овие особини го прават целењето кон митохондриската ДНК да е практична терапевтска стратегија.[72]
Биомаркери за рак[уреди | уреди извор]
Неколку биомаркери можат да бидат корисни во стадиумот, прогнозата и третманот на ракот. Тие може да варираат од еднонуклеотидни полиморфизми, хромозомски аберации, промени во бројот на копии на ДНК, нестабилност на микросателитите, метилација на промотерскиот регион, па дури и високи или ниски нивоа на белковини.[90] Помеѓу 2013 и 2019 година, само 6,8% од луѓето заболени од рак во две сојузни држави во Соединетите Држави биле подложени на генетско тестирање, што укажува на широко недоволно искористување на информациите што би можеле да ги подобрат одлуките за лекување и исходот на пациентите.[91]
Поврзано[уреди | уреди извор]
Наводи[уреди | уреди извор]
- ↑ 1,0 1,1 „Oncogenomics and the development of new cancer therapies“. Nature. 429 (6990): 469–74. мај 2004. Bibcode:2004Natur.429..469S. doi:10.1038/nature02627. PMID 15164073.
- ↑ Ting AH, McGarvey KM, Baylin SB (2006). „The cancer epigenome—components and functional correlates“. Genes Dev. 20 (23): 3215–31. doi:10.1101/gad.1464906. PMID 17158741.
- ↑ Jones PA, Baylin SB (2007). „The epigenomics of cancer“. Cell. 128 (4): 683–92. doi:10.1016/j.cell.2007.01.029. PMC 3894624. PMID 17320506.
- ↑ Li D, Zhang B, Xing X, Wang T (2015). „Combining MeDIP-seq and MRE-seq to investigate genome-wide CpG methylation“. Methods. 72: 29–40. doi:10.1016/j.ymeth.2014.10.032. PMC 4300244. PMID 25448294.
- ↑ Wei J, Li G, Dang S, Zhou Y, Zeng K, Liu M (2016). „Discovery and Validation of Hypermethylated Markers for Colorectal Cancer“. Dis. Markers. 2016: 2192853. doi:10.1155/2016/2192853. PMC 4963574. PMID 27493446.
- ↑ 6,0 6,1 „Whole-genome cancer analysis as an approach to deeper understanding of tumour biology“. Br. J. Cancer. 102 (2): 243–8. јануари 2010. doi:10.1038/sj.bjc.6605497. PMC 2816661. PMID 20029419.
- ↑ „Cancer genome landscapes“. Science. 339 (6127): 1546–58. 2013. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ↑ „Differences in DNA methylation signatures reveal multiple pathways of progression from adenoma to colorectal cancer“. Gastroenterology. 147 (2): 418–29.e8. 2014. doi:10.1053/j.gastro.2014.04.039. PMC 4107146. PMID 24793120.
- ↑ „DNA sequencing of a cytogenetically normal acute myeloid leukaemia genome“. Nature. 456 (7218): 66–72. ноември 2008. Bibcode:2008Natur.456...66L. doi:10.1038/nature07485. PMC 2603574. PMID 18987736.CS1-одржување: display-автори (link)
- ↑ 10,0 10,1 Peikoff, Kira (2015-10-16). „What Miraculous Recoveries Tell Us About Beating Cancer“. Popular Mechanics. Посетено на 20 февруари 2024.
- ↑ Bardelli A.; Velculescu V.E. (2005). „Mutational analysis of gene families in human cancer“. Current Opinion in Genetics & Development. 15 (1): 5–12. doi:10.1016/j.gde.2004.12.009. PMID 15661527.
- ↑ Benvenuti S.; Arena S.; Bardelli A. (2005). „Identification of cancer genes by mutational profiling of tumor genomes“. FEBS Letters. 579 (8): 1884–1890. doi:10.1016/j.febslet.2005.02.015. PMID 15763568.
- ↑ Shih I.M.; Wang T.L. (2005). „Apply innovative technologies to explore cancer genome“. Current Opinion in Oncology. 17 (1): 33–38. doi:10.1097/01.cco.0000147382.97085.e4. PMID 15608510.
- ↑ „1-Mb resolution array-based comparative genomic hybridization using a BAC clone set optimized for cancer gene analysis“. Genome Res. 14 (1): 179–87. јануари 2004. doi:10.1101/gr.1847304. PMC 314295. PMID 14672980.CS1-одржување: display-автори (link)
- ↑ „Representational oligonucleotide microarray analysis: a high-resolution method to detect genome copy number variation“. Genome Res. 13 (10): 2291–305. октомври 2003. doi:10.1101/gr.1349003. PMC 403708. PMID 12975311.CS1-одржување: display-автори (link)
- ↑ „Methylation-specific digital karyotyping“. Nat Protoc. 1 (3): 1621–36. 2006. doi:10.1038/nprot.2006.278. PMID 17406428.
- ↑ „Digital karyotyping reveals frequent inactivation of the dystrophin/DMD gene in malignant melanoma“. Cell Cycle. 6 (2): 189–98. јануари 2007. doi:10.4161/cc.6.2.3733. PMID 17314512.
- ↑ „End-sequence profiling: sequence-based analysis of aberrant genomes“. Proc. Natl. Acad. Sci. U.S.A. 100 (13): 7696–701. јуни 2003. Bibcode:2003PNAS..100.7696V. doi:10.1073/pnas.1232418100. PMC 164650. PMID 12788976.CS1-одржување: display-автори (link)
- ↑ Van, de Vijver M.J.; и др. (2002). „A gene-expression signature as a predictor of survival in breast cancer“. New England Journal of Medicine. 347 (25): 1999–2009. doi:10.1056/NEJMoa021967. PMID 12490681.
|hdl-access=бара|hdl=(help) - ↑ „Expression profiling predicts outcome in breast cancer“. Breast Cancer Res. 5 (1): 57–8. 2003. doi:10.1186/bcr562. PMC 154139. PMID 12559048.
- ↑ Xu Q.; Lee C. (2003). „Discovery of novel splice forms and functional analysis of cancer-specific alternative splicing in human expressed sequences“. Nucleic Acids Research. 31 (19): 5635–5643. doi:10.1093/nar/gkg786. PMC 206480. PMID 14500827.
- ↑ „Computational approaches to identify functional genetic variants in cancer genomes“. Nat. Methods. 10 (8): 723–9. август 2013. doi:10.1038/nmeth.2562. PMC 3919555. PMID 23900255.CS1-одржување: display-автори (link)
- ↑ „Operomics: molecular analysis of tissues from DNA to RNA to protein“. Clin. Chem. Lab. Med. 38 (9): 805–13. септември 2000. doi:10.1515/CCLM.2000.116. PMID 11097332.
- ↑ „Cross-species oncogenomics in cancer gene identification“. Cell. 125 (7): 1230–3. јуни 2006. doi:10.1016/j.cell.2006.06.018. PMID 16814709.
- ↑ „Mutational signatures and mutable motifs in cancer genomes“. Brief. Bioinformatics. 19 (6): 1085–1101. ноември 2018. doi:10.1093/bib/bbx049. PMC 6454500. PMID 28498882.
- ↑ „The clinical value of aberrant epigenetic changes of DNA damage repair genes in human cancer“. Oncotarget. 7 (24): 37331–37346. 2016. doi:10.18632/oncotarget.7949. PMC 5095080. PMID 26967246.
- ↑ „DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture“ (PDF). Mol. Cancer Res. 6 (4): 517–24. 2008. doi:10.1158/1541-7786.MCR-08-0020. PMID 18403632.
- ↑ Bernstein, C; Prasad, AR; Nfonsam, V; Bernstein, H. (2013). „Chapter 16: DNA Damage, DNA Repair and Cancer“. Во Chen, Clark (уред.). New Research Directions in DNA Repair. BoD – Books on Demand. стр. 413. ISBN 978-953-51-1114-6.
- ↑ „Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island“. PLOS Genetics. 4 (8): e1000155. 2008. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ↑ „DNA damage, homology-directed repair, and DNA methylation“. PLOS Genetics. 3 (7): e110. јули 2007. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ↑ „Molecular basis of base substitution hotspots in Escherichia coli“. Nature. 274 (5673): 775–80. август 1978. Bibcode:1978Natur.274..775C. doi:10.1038/274775a0. PMID 355893.
- ↑ „The CpG dinucleotide and human genetic disease“. Hum. Genet. 78 (2): 151–5. февруари 1988. doi:10.1007/bf00278187. PMID 3338800.
- ↑ 33,0 33,1 „Somatic hypermutagenesis in immunoglobulin genes. II. Influence of neighbouring base sequences on mutagenesis“. Biochim. Biophys. Acta. 1171 (1): 11–8. ноември 1992. doi:10.1016/0167-4781(92)90134-l. ISSN 0006-3002. PMID 1420357.
- ↑ „Somatic mutation hotspots correlate with DNA polymerase eta error spectrum“. Nat. Immunol. 2 (6): 530–6. јуни 2001. doi:10.1038/88732. ISSN 1529-2908. PMID 11376340.
- ↑ „Mutational processes molding the genomes of 21 breast cancers“. Cell. 149 (5): 979–93. мај 2012. doi:10.1016/j.cell.2012.04.024. PMC 3414841. PMID 22608084.CS1-одржување: display-автори (link)
- ↑ „Deciphering signatures of mutational processes operative in human cancer“. Cell Rep. 3 (1): 246–59. januari 2013. doi:10.1016/j.celrep.2012.12.008. PMC 3588146. PMID 23318258. Проверете ги датумските вредности во:
|date=(help) - ↑ „DeconstructSigs: delineating mutational processes in single tumors distinguishes DNA repair deficiencies and patterns of carcinoma evolution“. Genome Biol. 17: 31. февруари 2016. doi:10.1186/s13059-016-0893-4. PMC 4762164. PMID 26899170.
- ↑ „Exploring background mutational processes to decipher cancer genetic heterogeneity“. Nucleic Acids Res. 45 (W1): W514–W522. јули 2017. doi:10.1093/nar/gkx367. PMC 5793731. PMID 28472504.
- ↑ 39,0 39,1 39,2 „Passenger deletions generate therapeutic vulnerabilities in cancer“. Nature. 488 (7411): 337–42. август 2012. Bibcode:2012Natur.488..337M. doi:10.1038/nature11331. PMC 3712624. PMID 22895339.CS1-одржување: display-автори (link)
- ↑ „Collateral Lethality: A new therapeutic strategy in oncology“. Trends Cancer. 1 (3): 161–173. ноември 2015. doi:10.1016/j.trecan.2015.10.002. PMC 4746004. PMID 26870836.
- ↑ „Structure of the bis divalent cation complex with phosphonoacetohydroxamate at the active site of enolase“. Biochemistry. 31 (31): 7166–73. август 1992. doi:10.1021/bi00146a020. PMID 1322695.
- ↑ „Enolase activity and isoenzyme distribution in human brain regions and tumors“. J. Neurochem. 66 (6): 2484–90. јуни 1996. doi:10.1046/j.1471-4159.1996.66062484.x. PMID 8632173.
- ↑ 43,0 43,1 „SF2312 is a natural phosphonate inhibitor of enolase“. Nat. Chem. Biol. 12 (12): 1053–1058. декември 2016. doi:10.1038/nchembio.2195. PMC 5110371. PMID 27723749.CS1-одржување: display-автори (link)
- ↑ 44,0 44,1 „Genomic deletion of malic enzyme 2 confers collateral lethality in pancreatic cancer“. Nature. 542 (7639): 119–123. февруари 2017. Bibcode:2017Natur.542..119D. doi:10.1038/nature21052. PMC 5398413. PMID 28099419.CS1-одржување: display-автори (link)
- ↑ „Mutant KRas-Induced Mitochondrial Oxidative Stress in Acinar Cells Upregulates EGFR Signaling to Drive Formation of Pancreatic Precancerous Lesions“. Cell Rep. 14 (10): 2325–36. март 2016. doi:10.1016/j.celrep.2016.02.029. PMC 4794374. PMID 26947075.
- ↑ „A synthetic lethality-based strategy to treat cancers harboring a genetic deficiency in the chromatin remodeling factor BRG1“. Cancer Res. 73 (17): 5508–18. септември 2013. doi:10.1158/0008-5472.CAN-12-4593. PMID 23872584.CS1-одржување: display-автори (link)
- ↑ „Predictors and Modulators of Synthetic Lethality: An Update on PARP Inhibitors and Personalized Medicine“. Biomed Res Int. 2016: 2346585. 2016. doi:10.1155/2016/2346585. PMC 5013223. PMID 27642590.
- ↑ „RAD52 inactivation is synthetically lethal with deficiencies in BRCA1 and PALB2 in addition to BRCA2 through RAD51-mediated homologous recombination“. Oncogene. 32 (30): 3552–8. 2013. doi:10.1038/onc.2012.391. PMC 5730454. PMID 22964643.
- ↑ „Personalized synthetic lethality induced by targeting RAD52 in leukemias identified by gene mutation and expression profile“. Blood. 122 (7): 1293–304. 2013. doi:10.1182/blood-2013-05-501072. PMC 3744994. PMID 23836560.
- ↑ „Increased mutation rate at the hprt locus accompanies microsatellite instability in colon cancer“. Oncogene. 10 (1): 33–7. 1995. PMID 7824277.
- ↑ „PD-1 Blockade in Tumors with Mismatch-Repair Deficiency“. N. Engl. J. Med. 372 (26): 2509–20. 2015. doi:10.1056/NEJMoa1500596. PMC 4481136. PMID 26028255.CS1-одржување: display-автори (link)
- ↑ „SWI/SNF factors required for cellular resistance to DNA damage include ARID1A and ARID1B and show interdependent protein stability“. Cancer Res. 74 (9): 2465–75. 2014. doi:10.1158/0008-5472.CAN-13-3608. PMID 24788099.
- ↑ „Genome-Wide Transcriptional Regulation Mediated by Biochemically Distinct SWI/SNF Complexes“. PLOS Genet. 11 (12): e1005748. 2015. doi:10.1371/journal.pgen.1005748. PMC 4699898. PMID 26716708.
- ↑ „Discovery and saturation analysis of cancer genes across 21 tumour types“. Nature. 505 (7484): 495–501. 2014. Bibcode:2014Natur.505..495L. doi:10.1038/nature12912. PMC 4048962. PMID 24390350.
- ↑ „Promoter hypermethylation of ARID1A gene is responsible for its low mRNA expression in many invasive breast cancers“. PLOS ONE. 8 (1): e53931. 2013. Bibcode:2013PLoSO...853931Z. doi:10.1371/journal.pone.0053931. PMC 3549982. PMID 23349767.
- ↑ „ARID1A mutations in cancer: another epigenetic tumor suppressor?“. Cancer Discov. 3 (1): 35–43. 2013. doi:10.1158/2159-8290.CD-12-0361. PMC 3546152. PMID 23208470.
- ↑ „Synthetic lethality by targeting EZH2 methyltransferase activity in ARID1A-mutated cancers“. Nat. Med. 21 (3): 231–8. 2015. doi:10.1038/nm.3799. PMC 4352133. PMID 25686104.
- ↑ „SWI/SNF-mutant cancers depend on catalytic and non-catalytic activity of EZH2“. Nat. Med. 21 (12): 1491–6. 2015. doi:10.1038/nm.3968. PMC 4886303. PMID 26552009.
- ↑ „Synthetic Lethal Targeting of ARID1A-Mutant Ovarian Clear Cell Tumors with Dasatinib“ (PDF). Mol. Cancer Ther. 15 (7): 1472–84. 2016. doi:10.1158/1535-7163.MCT-15-0554. PMID 27364904.CS1-одржување: display-автори (link)
- ↑ Kaelin W.G. (2005). „The concept of synthetic lethality in the context of anticancer therapy“. Nature Reviews Cancer. 5 (9): 689–698. doi:10.1038/nrc1691. PMID 16110319.
- ↑ O'Connor M.J.; Martin N.M.B.; Smith G.C.M. (2007). „Targeted cancer therapies based on the inhibition of DNA strand break repair“. Oncogene. 26 (56): 7816–7824. doi:10.1038/sj.onc.1210879. PMID 18066095.
- ↑ „Targeting the DNA repair defect in BRCA mutant cells as a therapeutic strategy“. Nature. 434 (7035): 917–21. април 2005. Bibcode:2005Natur.434..917F. doi:10.1038/nature03445. PMID 15829967.
- ↑ Bryant H.E.; Schultz, Niklas; Thomas, Huw D.; Parker, Kayan M.; Flower, Dan; Lopez, Elena; Kyle, Suzanne; Meuth, Mark; Curtin, Nicola J.; и др. (2005). „Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase“. Nature. 434 (7035): 913–917. Bibcode:2005Natur.434..913B. doi:10.1038/nature03443. PMID 15829966.
- ↑ „IntOGen: integration and data mining of multidimensional oncogenomic data“. Nat. Methods. 7 (2): 92–3. февруари 2010. doi:10.1038/nmeth0210-92. PMID 20111033.
|hdl-access=бара|hdl=(help) - ↑ „IntOGen-mutations identifies cancer drivers across tumor types“. Nat. Methods. 10 (11): 1081–2. ноември 2013. doi:10.1038/nmeth.2642. PMC 5758042. PMID 24037244.
- ↑ „In silico prescription of anticancer drugs to cohorts of 28 tumor types reveals targeting opportunities“. Cancer Cell. 27 (3): 382–96. март 2015. doi:10.1016/j.ccell.2015.02.007. PMID 25759023.
|hdl-access=бара|hdl=(help) - ↑ „Oncogenic kinase signalling“. Nature. 411 (6835): 355–65. мај 2001. Bibcode:2001Natur.411..355B. doi:10.1038/35077225. PMID 11357143.
- ↑ „Mutational analysis of the tyrosine kinome in colorectal cancers“. Science. 300 (5621): 949. 2003. doi:10.1126/science.1082596. PMID 12738854.
- ↑ „High frequency of mutations of the PIK3CA gene in human cancers“. Science. 304 (5670): 554. април 2004. doi:10.1126/science.1096502. PMID 15016963.
- ↑ „Mutations of the BRAF gene in human cancer“ (PDF). Nature. 417 (6892): 949–54. јуни 2002. Bibcode:2002Natur.417..949D. doi:10.1038/nature00766. PMID 12068308.CS1-одржување: display-автори (link)
- ↑ Danson S.; Lorigan P. (2005). „Improving outcomes in advanced malignant melanoma – Update on systemic therapy“. Drugs. 65 (6): 733–743. doi:10.2165/00003495-200565060-00002. PMID 15819587.
- ↑ 72,0 72,1 72,2 72,3 72,4 Yu, Man (2012). Somatic Mitochondrial DNA Mutations in Human Cancers. Advances in Clinical Chemistry. 57. стр. 99–138. doi:10.1016/B978-0-12-394384-2.00004-8. ISBN 9780123943842. PMID 22870588.
- ↑ Hung, W.Y.; J.C. Lin; L.M. Lee; и др. (2008). „Tandem duplication/triplication correlated with poly-cytosine stretch variation in human mitochondrial DNA D-loop region“. Mutagenesis. 23 (2): 137–142. doi:10.1093/mutage/gen002. PMID 18252697.
- ↑ Fliss, M. S.; Usadel, H.; Caballero, O. L.; и др. (2000). „Facile detection of mitochondrial DNA mutations in tumors and bodily fluids“. Science. 287 (5460): 2017–2019. Bibcode:2000Sci...287.2017F. doi:10.1126/science.287.5460.2017. PMID 10720328.
- ↑ 75,0 75,1 Dani, M.A.; S.U. Dani; S.P. Lima; и др. (2004). „Less ΔmtDNA4977 than normal in various types of tumors suggests that cancer cells are essentially free of this mutation“. Genet. Mol. Res. 3 (3): 395–409. PMID 15614730.
- ↑ Ye, C.; X.O. Shu; W. Wen; и др. (2008). „Quantitative analysis of mitochondrial DNA 4977-bp deletion in sporadic breast cancer and benign breast diseases“. Breast Cancer Res. Treat. 108 (3): 427–434. doi:10.1007/s10549-007-9613-9. PMC 3836503. PMID 17541740.
- ↑ Tseng, L.M.; P.H. Yin; C.W. Chi; и др. (2006). „Mitochondrial DNA mutations and mitochondrial DNA depletion in breast cancer“. Genes Chromosomes Cancer. 45 (7): 629–638. doi:10.1002/gcc.20326. PMID 16568452.
- ↑ Zhu, W.; W. Qin; P. Bradley; A. Wessel; C.L. Puckett; E.R. Sauter (2005). „Mitochondrial DNA mutations in breast cancer tissue and in matched nipple aspirate fluid“. Carcinogenesis. 26 (1): 145–152. doi:10.1093/carcin/bgh282. PMID 15375011.
- ↑ „Frequency and phenotypic implications of mitochondrial DNA mutations in human squamous cell cancers of the head and neck“. Proc. Natl. Acad. Sci. U.S.A. 104 (18): 7540–5. мај 2007. Bibcode:2007PNAS..104.7540Z. doi:10.1073/pnas.0610818104. PMC 1863503. PMID 17456604.
- ↑ „Relationship between mitochondrial DNA instability, mitochondrial DNA large deletions, and nuclear microsatellite instability in head and neck squamous cell carcinomas“. Diagn. Mol. Pathol. 13 (1): 26–32. март 2004. doi:10.1097/00019606-200403000-00005. PMID 15163006.
- ↑ Tan, D.J.; J. Chang; W.L. Chen; и др. (2004). „Somatic mitochondrial DNA mutations in oral cancer of betel quid chewers“. Ann. N. Y. Acad. Sci. 1011 (1): 310–316. Bibcode:2004NYASA1011..310T. doi:10.1196/annals.1293.030. PMID 15126307.
- ↑ Lee, H.C.; S.H. Li; J.C. Lin; C.C. Wu; D.C. Yeh; Y.H. Wei (2004). „Somatic mutations in the D-loop and decrease in the copy number of mitochondrial DNA in human hepatocellular carcinoma“. Mutation Research. 547 (1–2): 71–78. doi:10.1016/j.mrfmmm.2003.12.011. PMID 15013701.
- ↑ Yin, P.H.; C.C. Wu; J.C. Lin; C.W. Chi; Y.H. Wei; H.C. Lee (2010). „Somatic mutations of mitochondrial genome in hepatocellular carcinoma“. Mitochondrion. 10 (2): 174–182. doi:10.1016/j.mito.2009.12.147. PMID 20006738.
- ↑ Tan, D.J.; J. Chang; L.L. Liu; и др. (2006). „Significance of somatic mutations and content alteration of mitochondrial DNA in esophageal cancer“. BMC Cancer. 6: 93. doi:10.1186/1471-2407-6-93. PMC 1459869. PMID 16620376.
- ↑ „Mitochondrial DNA mutations in pancreatic cancer“. Int J Gastrointest Cancer. 37 (2–3): 57–64. 2006. doi:10.1007/s12029-007-0008-2. PMID 17827523.
- ↑ „Somatic mutations in mitochondrial genome and their potential roles in the progression of human gastric cancer“. Biochim. Biophys. Acta. 1800 (3): 264–70. март 2010. doi:10.1016/j.bbagen.2009.06.006. PMID 19527772.
- ↑ „Mitochondrial DNA mutations and mitochondrial DNA depletion in gastric cancer“. Genes Chromosomes Cancer. 44 (1): 19–28. септември 2005. doi:10.1002/gcc.20213. PMID 15892105.
- ↑ Yu, J.J.; T. Yan (2010). „Effect of mtDNA mutation on tumor malignant degree in patients with prostate cancer“. Aging Male. 13 (3): 159–165. doi:10.3109/13685530903536668. PMID 20136572.
- ↑ Gomez-Zaera, M.; J. Abril; L. Gonzalez; и др. (2006). „Identification of somatic and germline mitochondrial DNA sequence variants in prostate cancer patients“. Mutation Research. 595 (1–2): 42–51. doi:10.1016/j.mrfmmm.2005.10.012. PMID 16472830.
- ↑ „Biomarkers in cancer staging, prognosis and treatment selection“. Nat. Rev. Cancer. 5 (11): 845–56. ноември 2005. doi:10.1038/nrc1739. PMID 16239904.
- ↑ Kurian (3 јули 2023). „Germline Genetic Testing After Cancer Diagnosis“. JAMA. 330 (1): 43–51. doi:10.1001/jama.2023.9526. PMC 10242510 Проверете ја вредноста
|pmc=(help). PMID 37276540 Проверете ја вредноста|pmid=(help). Посетено на 20 февруари 2024.
