Протеомика

Протеомика ― истражување на белковините на голем размер.[1][2] Белковините се витални делови на живите организми, со многу функции како што се образувањето на структурни влакна на мускулното ткиво, ензимско варење на храната или синтеза и репликација на ДНК. Покрај тоа, други видови на белковини вклучуваат антитела кои го штитат организмот од инфекции и хормони кои испраќаат важни сигнали низ телото.
Протеомот е целиот сет на белковинипроизведени или модифицирани од организам или систем. Протеомиката овозможува идентификација на постојано зголемување на бројот на белковини. Ова варира во зависност од времето и посебните барања, или стресови, под кои се подложува клетка или организам.[3]
Протеомиката е меѓудисциплинарен домен кој има голема корист од генетските информации на различни проекти за геном, вклучувајќи го и Проектот за човечкиот геном.[4] Го опфаќа истражувањето на протеомите од целокупното ниво на белковинскиот состав, структура и активност и е важна компонента на функционалната геномика.
Протеомиката воглавно означува опитна анализа од големи размери на белковини и протеоми, но често се однесува конкретно на прочистување на белковините и масена спектрометрија. Навистина, масената спектрометрија е најмоќниот метод за анализа на протеомите, и во големи примероци составени од милиони клетки[5] и во единечни клетки.[6][7]
Историја и етимологија[уреди | уреди извор]
Првите студии за белковини кои би можеле да се сметаат за протеомици започнале во 1975 година, по воведувањето на дводимензионалниот гел и мапирањето на белковини од бактеријата Escherichia coli.[се бара извор]
Зборот протеом е мешавина од зборовите „протеин“ (белковина) и „геном“. Тој бил измислен во 1994 година од тогашниот проф-др и студент Марк Вилкинс на Мекориевиот универзитет,[8] кој ја основал првата посветена лабораторија за протеомика во 1995 година.[9][10]
Сложеноста на проблемот[уреди | уреди извор]
По геномиката и транскриптомика, протеомиката е следниот чекор во проучувањето на биолошките системи. Тоа е посложено од геномиката бидејќи геномот на организмот е повеќе или помалку постојан, додека протеомите се разликуваат од клетка до клетка и од време на време. Различни гени се изразуваат во различни врсти на клетки, што значи дека дури и основниот сет на белковини произведени во клетката мора да биде идентификуван.
Во минатото овој феномен бил проценет со анализа на РНК, за која било откриено дека нема поврзаност со содржината на белковини.[11][12] Сега е познато дека информациската РНК не секогаш се преведува во белковина,[13] и количината на белковина произведен за дадена количина на информациската РНК зависи од генот од кој е транскрибиран и од физиолошката состојба на клетката. Протеомиката го потврдува присуството на белковината и дава директна мерка за неговата количина.
Послепреведувачки модификации[уреди | уреди извор]
Не само што преводот од информациската РНК предизвикува разлики, туку и многу белковини се подложени на широк спектар на хемиски модификации по преводот. Најчестите и најпроучени послепреведувачки модификации вклучуваат фосфорилација и гликозилација. Многу од овие послепреведувачки модификации се клучни за функцијата на белковината.
Фосфорилација[уреди | уреди извор]
Една таква модификација е фосфорилацијата, која им се случува на многу ензими и структурни белковини во постапката на клеточно сигнализирање. Додавањето на фосфат на одредени амино киселини - најчесто серин и треонин[14] со посредство на серин-треонински кинази, или поретко тирозин со посредство на тирозински кинази - предизвикува белковината да стане цел за врзување или меѓудејство со посебен сет на други белковината кои го препознаваат фосфорилираниот домен.
Бидејќи белковинската фосфорилација е една од најпроучените белковински модификации, многу „протеомски“ напори се насочени кон одредување на множеството фосфорилирани белковини во одредена клетка или ткивен вид под одредени околности. Ова го предупредува научникот за сигналните патишта кои можат да бидат активни во тој случај.
Убиквитинација[уреди | уреди извор]
Убикитин е мала белковина кој може да се прицврсти на одредени белковински супстрати со помош на ензими наречени Е3 убиквитински лигази. Утврдувањето кои белковини се поли-убиквитинираним помага да се разбере како се регулираат белковинските патишта. Според тоа, ова е дополнителна легитимна „протеомска“ студија. Слично на тоа, штом истражувачот ќе утврди кои супстрати се убиквитинирани од секоја лигаза, корисно е да се определи збирот на лигази изразени во одреден вид на клетка.[се бара извор]
Дополнителни модификации[уреди | уреди извор]
Покрај фосфорилацијата и убиквитинацијата, белковините може да бидат подложени на (меѓу другите) метилација, ацетилација, гликозилација, оксидација и нитрозилација. Некои белковини се подложени на сите овие модификации, често во комбинации зависни од времето. Ова ја илустрира потенцијалната сложеност на проучувањето на структурата и функцијата на белковините.
Различни белковини се направени под различни околности[уреди | уреди извор]
Клетката може да направи различни групи на белковини во различно време или под различни услови, на пример за време на развој, клеточна диференцијација, клеточен циклус или канцерогенеза. Понатамошно зголемување на сложеноста на протеомот, како што е споменато, повеќето белковини се способни да подлежат на широк опсег на послепреведувачки модификации.
Затоа, студијата за протеомиката може да стане сложена многу брзо, дури и ако темата на студијата е ограничена. Во поамбициозни услови, како на пример кога се бара биомаркер за посебен подвид на рак, научникот за протеомика може да избере да проучува повеќе примероци од крвен серум од повеќе пациенти со рак за да ги минимизира збунувачките фактори и да ја земе предвид опитна бучава.[15] Така, понекогаш се неопходни сложени опитни дизајни за да се земе предвид динамичната сложеност на протеомот.
Ограничувања на студиите за геномика и протеомика[уреди | уреди извор]
Протеомиката дава различно ниво на разбирање од геномиката поради многу причини:
- нивото на транскрипција на генот дава само груба проценка на неговото „ниво на превод“ во белковина.[16] Информациската РНК произведена во изобилство може брзо да биде разградувана или неефикасно да биде преведена, што резултира со мала количина на белковини.
- како што беше споменато погоре, многу белковини доживуваат „послепреведувачки модификации“ кои длабоко влијаат на нивните активности; на пример, некои белковини не се активни додека не се фосфорилираат. За проучување на послепреведувачките модификации се користени методи како што се фосфопротеомика и гликопротеомика.
- многу транскрипти доведуваат до повеќе од една белковина, преку алтернативно спојување или алтернативни послепреведувачки модификации.
- многу белковини образуваат комплекси со други белковини или молекули на РНК и функционираат само во присуство на овие други молекули.
- стапката на разградување на белковини игра важна улога во содржината на белковините.[17]
Можност за размножување
Еден главен фактор што влијае на можноста заразмножување во опитите со протеомика е истовременото елуирање на многу повеќе пептиди отколку што можат да измерат масените спектрометри. Ова предизвикува стохастички разлики помеѓу експериментите поради стекнувањето на триптични пептиди зависни од податоците. Иако раните големи протеомски анализи на пушка покажаа значителна променливост помеѓу лабораториите,[18][19] веројатно делумно поради техничките и опитните разлики помеѓу лабораториите, можноста за размножување е подобрена во поновата анализа на масена спектрометрија, особено на ниво на белковини.[20] Имено, насочената протеомика покажува зголемена можност за размножување и повторливост во споредба со методите со „сачмарка“, иако на сметка на густината и ефективноста на податоците.[21]
Квалитет на податоци
Протеомската анализа е многу подложна на автоматизација и се создаваат големи збирки податоци, кои се обработуваат со софтверски алгоритми. Параметрите на филтерот се користат за намалување на бројот на лажни погодоци, но тие не можат целосно да бидат елиминирани. Научниците ја изразиле потребата за свест дека протеомските опити треба да се придржуваат до критериумите на аналитичката хемија (доволен квалитет на податоците, проверка на разумност, валидација).[22][23][24][25]
Методи на проучување на белковините[уреди | уреди извор]
Во протеомиката, постојат повеќе методи за проучување на белковините. Општо земено, белковините може да бидат забележани со користење на антитела (имуноанализи), електрофоретско раздвојување или масена спектрометрија. Ако биде анализиран сложен биолошки примерок, или треба да биде користен многу специфично антитело во квантитативната анализа на точки, или биохемиското одвојување потоа треба да се користи пред чекорот на забележување, бидејќи има премногу аналити во примерокот за да се изврши точно откривање и квантификација.
Откривање на белковини со антитела (имуноанализа)[уреди | уреди извор]
Антителата на одредени белковини или на нивните модифицирани облици се користени во биохемиските и клеточните биолошки студии. Овие се меѓу најчестите алатки што ги користат молекуларните биолози денес. Постојат неколку специфични техники и протоколи кои користат антитела за откривање на белковини. Имуносорбентната анализа поврзана со ензимот е користена со децении за откривање и квантитативно мерење на белковините во примероците. Западниот Саутернов метод може да биде користен за откривање и квантификација на поединечни белковини, каде што во почетниот чекор, сложена белковинска смеса се одвојува со помош на SDS-PAGE, а потоа белковината од интерес се идентификува со користење на антитело.[се бара извор]
Модифицираните белковини може да бидат проучувани со развивање на антитело специфични за таа модификација. На пример, постојат антитела кои препознаваат одредени белковини само кога се тирозинско фосфорилирани, тие се познати како фосфо-специфични антитела. Исто така, постојат антитела специфични за други модификации. Тие може да бидат користени за да биде одреден збирот на белковини кои претрпеле модификација од интерес.
Имуноанализите може да се спроведат и со користење на рекомбинантно генерирани деривати на имуноглобулин или синтетички дизајнирани белковински скелиња кои се избрани за висока специфичност на антигенот. Таквите врзувачи вклучуваат фрагменти на антитела од еден домен (нанотела),[26] дизајнирани анкиринско повторувачки белковини[27] и аптамери.[28]
Откривањето на болеста на молекуларно ниво ја поттикнува новата револуција на рана дијагноза и третман. Предизвик со кој се соочува оваа област е дека белковинските биомаркери за рана дијагноза може да бидат присутни во многу мала изобилство. Долната граница на откривање со конвенционалната технологија за имуноанализа е горниот фемтомоларен опсег (10−13 М). Технологијата за дигитална имуноанализа ја подобри чувствителноста на откривање на три логови, до атомоларниот опсег (10−16 М). Оваа способност има потенцијал да отвори нови достигнувања во дијагностиката и терапевтската медицина, но таквите технологии се префрлени на рачни постапки кои не се добро прилагодени за ефикасна рутинска употреба.[29]
Откривање на белковини без антитела[уреди | уреди извор]
Додека откривањето на белковини со антитела е сè уште многу честа појава во молекуларната биологија, развиени се и други методи кои не се потпираат на антитела. Овие методи нудат различни предности, на пример тие често се способни да ја одредат секвенцата на белковината или пептидот, тие може да имаат поголема пропусност од онаа заснована на антитела, и понекогаш можат да ги идентификуваат и квантифицираат белковините за кои не постојат антитела.
Методи за откривање[уреди | уреди извор]
Еден од најраните методи за анализа на белковините е Едмановото разградување (воведена во 1967 година) каде што еден пептид е подложен на повеќе чекори на хемиска разградување за да биде разрешена неговата низа. Овие рани методи се претежно заменети со технологии кои нудат поголема пропусност.[се бара извор]
Неодамна применетите методи користат техники засновани на масена спектрометрија, развој што бил овозможен со откривањето на методите за „мека јонизација“ развиени во 1980-тите, како што се ласерска десорпција/јонизација со помош на матрица и јонизација со електропрскање. Овие методи довеле до појава на работни текови на протеомика од горе-надолу и одоздола-нагоре каде што често се врши дополнително раздвојување пред анализата (видете подолу).
Методи на раздвојување[уреди | уреди извор]
За анализа на сложени биолошки примероци, потребно е намалување на сложеноста на примерокот. Ова може да се изврши вонмрежно со еднодимензионално или дводимензионално одвојување. Во поново време, семрежните методи се развиени каде што поединечни пептиди (во пристапите на протеомика одоздола нагоре) се одвојуваат со помош на хроматографија во обратна фаза, а потоа директно се јонизираат со помош на јонизација со електропрскање; директното спојување на одвојувањето и анализата го објаснува поимот „семрежна“ анализа.
Хибридни технологии[уреди | уреди извор]
Постојат неколку хибридни технологии кои користат прочистување врз основа на антитела на поединечни аналити и потоа вршат масена спектрометриска анализа за идентификација и квантификација. Примери за овие методи се MSIA (массовен спектрометриски имуноанализа), развиен од Рандал Нелсон во 1995 година,[30] и методот SISCAPA (Стабилен изотопски стандарден фаќање со антипептидни антитела), воведен од Ли Андерсон во 2004 година.[31]
Тековни методологии на истражување[уреди | уреди извор]
Флуоресцентна дводимензионална диференцијална гелна електрофореза (2-D DIGE)[32] може да биде користена за квантифицирање на варијациите во постапката 2-D DIGE и воспоставување статистички валидни прагови за доделување квантитативни промени помеѓу примероците.[32]
Компаративната протеомска анализа може да ја открие улогата на белковините во сложените биолошки системи, вклучувајќи го и размножувањето. На пример, третманот со инсектицидот триазофос предизвикува зголемување на содржината на кафеави растителни жлезди (Nilaparvata lugens (Стол)) белковини на машките помошни жлезди кои може да бидат пренесени на женките преку парење, предизвикувајќи зголемување на плодноста (т.е. стапка на наталитет) на женките.[33] За да се идентификуваат промените во врстите на белковините на придружните жлезди (Acps) и репродуктивните белковини кои ги спарувале женските жлезди кои ги добиле од машките жлезди, истражувачите спроведоа споредбена протеомска анализа на спарените женки N. lugens.[34] Резултатите покажале дека овие белковини учествуваат во репродуктивната постапка на возрасните женски и машки единки на N. lugens.[34]
Протеомската анализа на Arabidopsis peroxisomes [35] е воспоставена како главен непристрасен пристап за идентификација на нови пероксизомални белковини во голем обем.[35]
Постојат многу пристапи за карактеризирање на човечкиот протеом, кој се проценува дека содржи помеѓу 20.000 и 25.000 не-излишни белковини. Бројот на уникатни белковински видови најверојатно ќе се зголеми за помеѓу 50.000 и 500.000 поради настаните на спојување и протеолиза на РНК, а кога ќе се разгледаат и послепреведувачката модификација, се проценува дека вкупниот број на уникатни човечки белковини се движи во ниски милиони.[36][37]
Покрај тоа, неодамна биле пријавени првите ветувачки обиди за дешифрирање на протеомот на животинските тумори.[38] Овој метод бил користен како функционален метод во профилирањето на белковините на Macrobrachium rosenbergii.[39]
Протеомски технологии со висока пропусност[уреди | уреди извор]
Протеомиката постојано добива на интензитет во текот на изминатата деценија со еволуцијата на неколку пристапи. Неколку од нив се нови, а други се надоврзуваат на традиционални методи. Методите засновани на масена спектрометрија, протеомиката на афинитет и микро низите се најчестите технологии за големо проучување на белковините.
Спектрометрија на маса и профилирање на белковини[уреди | уреди извор]

Моментално се користат два методи засновани на масена спектрометрија за профилирање на белковини. Повоспоставениот и широко распространет метод користи висока резолуција, дводимензионална електрофореза за паралелно одвојување на белковините од различни примероци, проследено со избор и боење на диференцијално изразените белковини кои треба да се идентификуваат со масена спектрометрија. И покрај напредокот во 2-DE и неговата зрелост, тој има и свои граници. Централната грижа е неможноста да се разрешат сите белковини во примерокот, со оглед на нивниот драматичен опсег во нивото на изразување и различните својства. Комбинацијата на големината на порите и полнењето на белковините, големината и обликот може во голема мера да ја одреди стапката на селење што доведува до други усложувања.[40]
Вториот квантитативен пристап користи стабилни изотопски ознаки за диференцијално означување на белковините од две различни сложени мешавини.[41][42] Овде, белковините во сложената смеса прво се означени изотопски, а потоа се вари за да бидат добиени означени пептиди. Обележаните мешавини потоа се комбинирани, пептидите се одделувани со повеќедимензионална течна хроматографија и се анализирани со тандемска масена спектрометрија. Реагенсите за афинитет со кодирани изотопски ознаки (ICAT) се широко користени изотопски ознаки. Во овој метод, цистеинските остатоци од белковините ковалентно се прикачуваат на ICAT реагенсот, со што се намалува сложеноста на мешавините испуштајќи ги не-цистеинските остатоци.
Квантитативната протеомика која користи стабилно изотопско обележување е сè покорисна алатка во современиот развој. Прво, хемиските реакции се користени за воведување ознаки во одредени места или белковини со цел да бидат испитани специфичните функционалности на белковините. Изолацијата на фосфорилирани пептиди е постигната со користење на изотопско означување и избрани хемикалии за да се долови фракцијата на белковината меѓу сложената смеса. Второ, технологијата ICAT била искористена за да биде направена разлика помеѓу делумно прочистените или прочистените макромолекуларни комплекси како што се големиот комплекс на РНК полимераза II пред иницијација и белковината сложени со фактор на транскрипција на квасец. Трето, означувањето на ICAT неодамна било комбинирано со изолација на хроматин за да се идентификуваат и квантифицираат белковините поврзани со хроматин. Конечно, ICAT реагенсите се корисни за протеомско профилирање на клеточните органели и специфичните клеточни фракции.[40]
Друг квантитативен пристап е пристапот со точни ознаки за маса и време развиен од Ричард Д. Смит и соработниците во Тихоокеанската северозападна национална лабораторија. Во овој пристап, зголемена пропусност и чувствителност се постигнувани со избегнување на потребата за тандемска масена спектрометрија и користење на прецизно определени информации за времето на одделување и високо точни определувања на масата за идентификација на пептиди и белковини.
Афинитетна протеомика[уреди | уреди извор]
Афинитетната протеомика користи антитела или други афинитетни реагенси (како што се аптамери засновани на олигонуклеотиди) како белковински специфични сонди за откривање.[43] Во моментов овој метод може да испита неколку илјади белковини, обично од биофлуиди како што се плазма, серум или цереброспинална течност. Клучна разлика за оваа технологија е способноста да бидат анализирани стотици или илјадници примероци во разумна временска рамка (прашање на денови или недели); Методите засновани на масена спектрометрија не се скалабилни до ова ниво на пропусност на примерокот за протеомски анализи.
Белковински чипови[уреди | уреди извор]
Урамнотежувањето на употребата на масени спектрометри во протеомиката и во медицината е употребата на белковински микро низи. Целта зад белковинските микро низи е да се испечатат илјадници одлики за откривање белковини за испрашување на биолошки примероци. Низите на антитела се пример во кој мноштво различни антитела се наредени за откривање на нивните соодветни антигени од примерок од човечка крв. Друг пристап е распоредувањето на повеќе видови белковини за проучување на својствата како меѓудејствија со белковина-ДНК, белковина-белковинаи белковина-лиганд. Идеално, функционалните протеомски низи би го содржат целиот комплемент од белковините на даден организам. Првата верзија на таквите низи се состоеше од 5000 прочистени белковини од квасец депонирани на стаклени микроскопски слајдови. И покрај успехот на првиот чип, беше поголем предизвик да бидат применети белковински низи. Белковините се инхерентно многу потешки за работа од ДНК. Тие имаат широк динамички опсег, се помалку стабилни од ДНК и нивната структура е тешко да се зачува на стаклени слајдови, иако тие се од суштинско значење за повеќето анализи. Светската ICAT технологија има впечатливи предности во однос на технологиите за белковински чипови.[40]
Белковински микронизи во обратна фаза[уреди | уреди извор]
Ова е ветувачка и понова принема за микросреди за дијагноза, проучување и третман на сложени болести како што е ракот. Технологијата ја спојува микродисекцијата за фотографирање со ласер со технологијата на микро низа, за да произведе белковински микронизи во обратна фаза. Во овој тип на микросреди, целата збирка на белковини се имобилизирани со намера да бидат фатени различни фази на болеста кај поединечен пациент. Кога се користени со микродисекцијата, низите со обратна фаза можат да ја следат флуктуирачката состојба на протеомот кај различно клеточно население во мала област од човечкото ткиво. Ова е корисно за профилирање на статусот на клеточните сигнални молекули, меѓу пресекот на ткивото што вклучува и нормални и канцерогени клетки. Овој пристап е корисен за следење на статусот на клучните фактори во нормалниот епител на простата и инвазивните ткива на рак на простата. LCM потоа ги сецира овие ткивни и белковинските лизати биле наредени на нитроцелулозни слајдови, кои биле испитани со специфични антитела. Овој метод може да ги следи сите видови на молекуларни настани и може да ги споредува заболените и здравите ткива во ист пациент што овозможува развој на стратегии за третман и дијагноза. Способноста да се добијат протеомични снимки од соседните клеточни населенија, со користење на микронизи со обратна фаза во врска со микродисекцијата има голем број примени надвор од проучувањето на туморите. Пристапот може да обезбеди увид во нормалната физиологија и патологија на сите ткива и е непроценлив за карактеризирање на развојните постапки и аномалии.[40]
Откривање на белковини преку биоортогонална хемија[уреди | уреди извор]
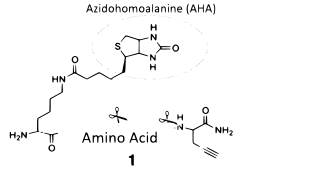
Неодамнешните достигнувања во биоортогоналната хемија откриле примени во анализата на белковините. Проширувањето на користењето органски молекули за да биде набљудувана нивната реакција со белковините открива широки методи за нивно обележување. Неприродните амино киселини и различните функционални групи претставуваат нови растечки технологии во протеомиката.
Специфичните биомолекули кои се способни да се метаболизираат во клетките или ткивата се вметнуваат во белковини или гликани. Молекулата ќе има ознака за афинитет, модифицирајќи ја белковината што овозможува негово откривање. Азидохомоаланин (АХА) ја користи оваа ознака за афинитет преку вклучување со Met-t-RNA синтетаза за да се вклучување во белковините. Ова му овозможило на АХА да помогне во одредувањето на идентитетот на новосинтетизираните белковини создадени како одговор на пертурбациите и да ги идентификува белковините што се секретираат од клетките.[44]
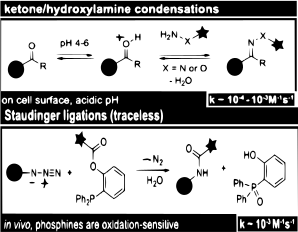
Неодамнешните студии[45] користејќи кондензации на кетони и алдехиди покажуваат дека тие се најпогодни за ин витро или означување на клеточната површина. Сепак, користењето на кетони и алдехиди како биоортогонални известувачи открила бавна кинетика што укажува дека иако е ефикасно за означување, концентрацијата мора да биде висока.
Одредени белковини може да бидат забележани преку нивната реактивност на азидни групи . Небелковинородните амино киселини можат да носат азидни групи кои реагираат со фосфини во Стаудингеровите лигации. Оваа реакција веќе е искористена за означување на други биомолекули во живите клетки и животните.[46]
Биоортоганското поле се шири и поттикнува дополнителни примени во рамките на протеомиката. Вреди да се забележат ограничувањата и придобивките. Брзите реакции можат да создадат биоконјукции и да создадат високи концентрации со мали количини на реактанти. Спротивно на бавните кинетички реакции како кондензација на алдехид и кетон, додека се ефективни, бараат висока концентрација што го прави неефикасен.
Практични примени[уреди | уреди извор]
Ново откритие за лекови[уреди | уреди извор]
Еден главен развој што доаѓа од проучувањето на човечките гени и белковини е идентификацијата на потенцијални нови лекови за третман на болести. Ова се потпира на информации за геномот и протеомот за да бидат идентификувани белковините поврзани со болеста, кои сметачки софтвер потоа може да ги користи како цели за нови лекови. На пример, ако одредена белковина е вмешана во некоја болест, неговата тродимензионална структура обезбедува информации за дизајнирање на лекови кои ќе го попречат дејството на белковината. Молекулата која одговара на активното место на ензимот, но не може да се ослободи од ензимот, го исклучува ензимот. Ова е основата на новите алатки за откривање лекови, кои имаат за цел да најдат нови лекови за инактивирање на белковините вклучени во болеста. Како што се наоѓаат генетските разлики меѓу поединците, истражувачите очекуваат да ги користат овие техники за да развијат персонализирани лекови кои се поефикасни за поединецот.[47]
Протеомиката исто така е користена за откривање на сложени меѓудејствија помеѓу растенијата и инсектите кои помагаат да бидат идентификувани кандидатските гени вклучени во одбранбениот одговор на растенијата кон тревопасните животни.[48][49][50]
Огранок на протеомиката наречена хемопротеомика обезбедува бројни алатки и техники за откривање на белковински цели на лековите.[51]
Меѓудејствувачка протеомика и белковински мрежи[уреди | уреди извор]
Меѓудејствувачката протеомика е анализа на белковинските меѓудејствија од размери на бинарни меѓудејствија до протеомски или ширум мрежата. Повеќето белковини функционираат преку меѓудејствија на белковина со белковина, а една цел на меѓудејствувачката протеомика е да бидат идентификуваат бинарни белковински меѓудејствија, белковински комплекси и интерактоми.
Достапни се неколку методи за испитување на меѓудејствијата белковина со белковина. Додека најтрадиционалниот метод е двохибридна анализа на квасец, моќен метод што се појавува е афинитетното прочистување проследено со белковина масена спектрометрија користејќи означени белковински мамки. Други методи вклучуваат површинска плазмонска резонанца,[52][53] белковински микронизи, интерферометрија со двојна поларизација, термофореза во микроскала, анализа на кинетичко исклучување и опитни методи како што се прикажување на бактериофаг и сметачки методи „in silico“.
Познавањето за меѓудејствијата помеѓу белковини со белковини е особено корисно во однос на биологијата на биолошките мрежи и системи, на пример во каскадите за сигнализирање на клетките и регулаторните мрежи на гените (каде што знаењето за меѓудејствијата помеѓу белковините и ДНК е исто така информативно). Анализата на белковинските меѓудејствија на ниво на протеом и интеграцијата на овие модели на меѓудејствие во поголеми биолошки мрежи е од клучно значење за разбирање на биологијата на ниво на систем.[54][55]
Изразувачка протеомика[уреди | уреди извор]
Изразувачката протеомика вклучува анализа на белковинското изразување во поголем обем. Тоа помага да бидат идентификувани главните белковини во одреден примерок, и оние белковини диференцијално изразени во сродни примероци - како што е заболеното наспроти здравото ткиво. Ако белковината е најдена само во заболен примерок, тогаш таа може да биде корисна цел на лекот или дијагностички маркер. Белковините со исти или слични профили на изразување исто така може да бидат функционално поврзани. Постојат технологии како 2D-PAGE и масена спектрометрија кои се користени во изразната протеомика.[56]
Биомаркери[уреди | уреди извор]
Националниот институт за здравје (од Соединетите Држави) го дефинира биомаркерот како „одлика која објективно се мери и оценува како показател за нормални биолошки постапки, патогени постапки или фармаколошки одговори на терапевтска интервенција“.[57][58]
Разбирањето на протеомот, структурата и функцијата на секоја белковина и сложеноста на меѓудејствијата на белковина со белковина се од клучно значење за развивање на најефективните дијагностички техники и третмани на болести во иднина. На пример, протеомиката е многу корисна во идентификацијата на кандидатите биомаркери (белковини во телесните течности кои се важни за дијагноза), идентификација на бактериски антигени кои се насочени од имунолошкиот одговор и идентификација на можни имунохистохемиски маркери на заразни или неопластични болести.[59]
Интересна употреба на протеомика е користење на специфични белковински биомаркери за дијагностицирање на болеста. Голем број техники овозможуваат тестирање на белковини произведени за време на одредена болест, што помага брзо да се дијагностицира болеста. Техниките вклучуваат западен Саутернов метод, имунохистохемиско боење, ензимско поврзан имуносорбентен тест или масена спектрометрија.[38][60] Секретомиката, подобласт на протеомика која ги проучува секретираните белковини и патиштата на секреција користејќи протеомски пристапи, неодамна се појавила како важна алатка за откривање на биомаркери на болеста.[61]
Протеогеномика[уреди | уреди извор]
Во протеогеномиката, протеомските технологии како што е масената спектрометрија се користени за подобрување на генските прибелешки. Напоредната анализа на геномот и протеомот го олеснува откривањето на послепреведувачки модификации и протеолитички настани,[62] особено кога се споредуваат повеќе видови (споредбена протеогеномика).[63]
Структурна протеомика[уреди | уреди извор]
Структурната протеомика вклучува анализа на белковинска структури во големи размери. Ги споредува белковинската структури и помага да бидат идентификувани функциите на новооткриените гени. Структурната анализа, исто така, помага да биде разбрана дека каде лековите се врзуваат за белковините и исто така покажува каде белковините општат едни со други. Ова разбирање се постигнува со користење на различни технологии како што се рендгенска кристалографија и NMR спектроскопија.
Биоинформатика за протеомика (протеомска информатика)[уреди | уреди извор]
Многу податоци за протеомиката се собирани со помош на технологии со висока пропусна моќ, како што се масена спектрометрија и микронизи. Честопати се потребни недели или месеци за да бидат анализирани податоците и да бидат направени споредби рачно. Поради оваа причина, биолозите и хемичарите соработуваат со информатичари и математичари за да создадат програми и обработени податоци за пресметковна анализа на белковинските податоци. Користејќи биоинформатички техники, истражувачите се способни за побрза анализа и складирање на податоци. Добро место за наоѓање списоци со тековни програми и бази на податоци е на порталот за биоинформатички ресурси ExPASy. Примените на протеомика заснована на биоинформатика вклучуваат медицина, дијагноза на болести, идентификација на биомаркери и многу повеќе.
Идентификација на белковини[уреди | уреди извор]
Масовната спектрометрија и микронизата произведуваат информации за фрагментација на пептиди, но не даваат идентификација на специфични белковини присутни во оригиналниот примерок. Поради недостаток на специфична идентификација на белковините, минатите истражувачи биле принудени самите да ги дешифрираат фрагментите на пептидот. Сепак, во моментов има достапни програми за идентификација на белковини. Овие програми ги земаат пептидните секвенци од масената спектрометрија и микронизата и враќаат информации за соодветни или слични белковини. Ова се прави преку алгоритми применети од програмата кои вршат порамнување со белковини од познати бази на податоци како што се UniProt[64] и PROSITE[65] за да биде предвидено кои белковини се во примерокот со одреден степен на сигурност.
Структура на белковини[уреди | уреди извор]
Биомолекуларната структура ја образува тродимензионалната конфигурација на белковината. Разбирањето на структурата на белковината помага во идентификацијата на меѓудејствијата и функцијата на белковината. Порано било дека тродимензионалната структура на белковините можела да биде одредена само со помош на рендгенска кристалографија и NMR спектроскопија. Од 2017 година, крио-електронската микроскопија е водечка техника, која ги решава тешкотиите со кристализација (во рендгенската кристалографија) и конформациската двосмисленост (во NMR); резолуцијата била 2,2 А од 2015 година. Сега, преку биоинформатиката, постојат сметачки програми кои во некои случаи можат да ја предвидат и моделираат структурата на белковините. Овие програми ги користат хемиските својства на аминокиселините и структурните својства на познатите белковини за да го предвидат 3Д моделот на примероци од белковини. Ова исто така им овозможува на научниците да ги моделираат белковинските меѓудејствија во поголем обем. Покрај тоа, биомедицинските инженери развиваат методи за фактор во флексибилноста на белковинските структури за да прават споредби и предвидувања.[66]
Послепреведувачки модификации[уреди | уреди извор]
Повеќето програми достапни за анализа на белковини не се напишани за белковини кои претрпеле послепреведувачки модификации.[67] Некои програми ќе прифатат послепреведувачки модификации за да помогнат во идентификацијата на белковините, но потоа ќе ја игнорираат модификацијата за време на понатамошната анализа на белковините. Важно е да се земат предвид овие модификации бидејќи тие можат да влијаат на структурата на белковината. За возврат, пресметковната анализа на послепреведувачките модификации го привлече вниманието на научната заедница. Тековните послепреведувачки програми за модификација се само предвидливи.[68] Хемичари, биолози и информатичари работат заедно за да создадат и воведат нови податочно обработени низи кои овозможуваат анализа на послепреведувачки модификации кои се опитно идентификувани за нивниот ефект врз структурата и функцијата на белковината.
Сметачки методи во проучувањето на белковинските биомаркери[уреди | уреди извор]
Еден пример за употреба на биоинформатиката и употреба на сметачки методи е проучувањето на белковинските биомаркери. Пресметувачките предвидувачки модели[69] покажале дека обемната и разновидна размена со фето-мајчини белковини се јавува за време на бременоста и може лесно да се открие неинвазивно во целата мајчина крв. Овој пресметковен пристап заобиколи големо ограничување, изобилството на мајчини белковини кои се мешаат во откривањето на феталните белковини, до феталната протеомска анализа на мајчината крв. Пресметковните модели можат да користат транскрипти на фетални гени претходно идентификувани во целата крв на мајката за да создадат сеопфатна протеомска мрежа на поимотновороденче. Таквата работа покажува дека феталните белковини откриени во крвта на бремената жена потекнуваат од разновидна група на ткива и органи од фетусот во развој. Протеомските мрежи содржат многу биомаркери кои се прокси за развој и ја илустрираат потенцијалната клиничка примена на оваа технологија како начин за следење на нормалниот и абнормалниот развој на фетусот.
Воведена е и информациско-теоретска рамка за откривање на биомаркери, интегрирајќи информации за биофлуид и ткиво.[70] Овој нов пристап ја користи функционалната синергија помеѓу одредени биофлуиди и ткива со потенцијал за клинички значајни наоди што не се можни доколку ткивата и биофлуидите се разгледуваат поединечно. Со концептуализирање на ткивниот биофлуид како информациски канали, може да се идентификуваат значајни биофлуидни посредници и потоа да бидат користени за воден развој на клиничка дијагностика. Кандидатите биомаркери потоа се предвидувани врз основа на критериумите за пренос на информации низ каналите на ткиво-биофлуид. Значајни односи биофлуид-ткиво може да бидат користени за да биде даден приоритет на клиничката валидација на биомаркерите.[70]
Нови трендови[уреди | уреди извор]
Голем број на концепти кои се појавуваат имаат потенцијал да ги подобрат сегашните одлики на протеомиката. Добивањето апсолутна квантификација на белковините и следењето на послепреведувачките модификации се двете задачи кои влијаат на разбирањето на белковинската функција кај здравите и заболените клетки. За многу клеточни настани, концентрациите на белковините не се менуваат; туку нивната функција е модулирана со послепреведувачки модификации. Методите за следење на модификациите се недоволно развиена област во протеомиката. Изборот на одредена подгрупа на белковина за анализа значително ја намалува сложеноста на белковината, што ја прави поволен за дијагностички цели каде што крвта е почетен материјал. Друго важно гледиште на протеомиката, кое сè уште не е обработено, е дека методите на протеомика треба да бидат насочени на проучување на белковините во контекст на животната средина. Зголемената употреба на хемиски вкрстени поврзувачи, воведени во живите клетки за да се поправат белковина со белковина, белковина со ДНК и други меѓудејствија, може делумно да го подобри овој проблем. Предизвикот е да бидат идентификувани соодветни методи за зачувување на релевантните меѓудејствија. Друга цел за проучување на белковините е да бидат развиени пософистицирани методи за сликање на белковините и другите молекули во живите клетки и во вистинско време.[40]
Системска биологија[уреди | уреди извор]
Напредокот во квантитативната протеомика јасно ќе овозможи подлабока анализа на клеточните системи.[54][55] Друга истражувачка граница е анализата на единечни клетки,[71][72] и белковинската ковариција низ единечни клетки[73] што ги одразува биолошките постапки како што се образување на белковински комплекси, имунолошки функции,[74] како и клеточен циклус и подготвување на ракот клетки за отпорност на лекови.[75] Биолошките системи се предмет на различни пертурбации (клеточен циклус, клеточна диференцијација, канцерогенеза, животна средина (биофизичка) итн.). Транскрипциските и транслационите одговори на овие пертурбации резултираат со функционални промени на протеомот вмешан како одговор на стимулот. Затоа, опишувањето и квантифицирањето на промените на протеомот во изобилството на белковини е од клучно значење за похолистичко разбирање на биолошкиот феномен, на ниво на целиот систем. На овој начин, протеомиката може да се гледа како комплементарна на геномиката, транскриптомиката, епигеномиката, метаболомиката и други гранки со завршетокот „-омика“ во интегративните анализи кои се обидуваат да ги дефинираат биолошките фенотипови посеопфатно. Како пример, The Cancer Proteome Atlas обезбедува квантитативни податоци за изразување на белковини за ~200 белковини во над 4.000 примероци на тумор со усогласени транскриптомски и геномски податоци од The Cancer Genome Atlas.[76] Слични сетови на податоци во други врсти клетки, врсти ткива и видови, особено со користење на длабока масена спектрометрија, ќе бидат неизмерно важен ресурс за истражување во области како биологија на ракот, развојна и биологија на матични клетки, медицина и еволутивна биологија.
Протеом на човечка плазма[уреди | уреди извор]
Карактеризирањето на протеомот на човечката плазма станало главна цел во протеомската арена, но исто така е и најпредизвикувачкиот протеом од сите човечки ткива.[77] Содржи имуноглобулин, цитокини, белковински хормони и секретирани белковини кои укажуваат на инфекција над резидентни, хемостатични белковини. Исто така, содржи белковини кои протекуваат ткиво поради циркулацијата на крвта низ различни ткива во телото. Така, крвта содржи информации за физиолошката состојба на сите ткива и, во комбинација со неговата пристапност, го прави крвниот протеом непроценлив за медицински цели. Се смета дека карактеризирањето на протеомот на крвната плазма е застрашувачки предизвик.
Длабочината на плазма протеомот опфаќа динамичен опсег од повеќе од 1010 помеѓу највисоката изобилна белковина (албумин) и најнискиот (некои цитокини) и се смета дека е еден од главните предизвици за протеомиката.[78] Временската и просторната динамика дополнително го усложуваат проучувањето на протеомот на човечката плазма. Протокот на некои белковини е доста побрз од другите и содржината на белковини во артеријата може значително да се разликува од онаа на вената. Сите овие разлики прават дури и наједноставната протеомска задача за каталогизација на протеомот да изгледа недостапна. За да биде решен овој проблем, треба да бидат утврдени приоритети. Фаќањето на најзначајната подгрупа на белковини меѓу целиот протеом за да биде створена дијагностичка алатка е еден таков приоритет. Второ, бидејќи ракот е поврзан со зголемена гликозилација на белковините, методите што се насочуваат на овој дел од белковините исто така ќе бидат корисни. Повторно: повеќепараметарската анализа најдобро открива патолошка состојба. Како што се подобруваат овие технологии, профилите на болеста треба постојано да бидат поврзани со соодветните промени во генското изразување.[40] Поради горенаведените проблеми, протеомијата на плазмата останала предизвик. Сепак, технолошкиот напредок и постојаниот развој се чини дека резултираат со оживување на плазма протеомиката како што било покажано неодамна со технологијата наречена профилирање на плазмен протеом.[79] Благодарение на таквите технологии, истражувачите биле во можност да ги истражат постапките на воспаление кај глувците, наследноста на плазмените протеоми, како и да го покажат ефектот од таквата вообичаена промена на животниот стил како што е губење на тежината на плазмениот протеом.[80][81][82]
Списанија[уреди | уреди извор]
Бројни списанија се посветени на полето на протеомиката и сродните области. Забележете дека списанијата што се занимаваат со белковини обично се повеќе насочени на структурата и функцијата, додека списанијата за протеомика се повеќе насочени на анализа од големи размери на цели протеоми или барем големи групи на белковини. Некои од поважните се наведени подолу (со нивните издавачи).
- Molecular and Cellular Proteomics (ASBMB)
- Journal of Proteome Research (ACS)
- Journal of Proteomics (Elsevier)
- Proteomics (Wiley)
Поврзано[уреди | уреди извор]
- Протеомика заснована на активност
- Превртена протеомика
- Цитомика
- Функционална геномика
- Топлинска стабилизација
- Проект за човековиот протеом
- Имунопротеомика
- Список на биолошки податочни бази
- Список на теми за омика во биологијата
- Пегилација
- Фосфопротеомика
- Белковинско производство
- Протеогеномика
- Претеомска хемија
- Секретомика
- Сачмарска протеомика
- Исправена протеомика
- Системска биологија
- Двохибриден преглед
- Преведено секвенционирање на сложен профил
- Гликомика
Белковински бази на податоци[уреди | уреди извор]
- Атлас на човековата белковина
- Референтна податочна база на човековата белковина
- Национален центар за биотехнолошки информации
- ПептидАтлас
- Банка на податоци за белковини
- Информациски ресурс за белковини
- Податочна база за индентификации за протеомиката
- Протеопедија—соработничка, тродимензионална енциклопедија на белковини и други молекули
- УниПрот
Истражувачки центри[уреди | уреди извор]
Наводи[уреди | уреди извор]
- ↑ „Proteome and proteomics: new technologies, new concepts, and new words“. Electrophoresis. 19 (11): 1853–1861. август 1998. doi:10.1002/elps.1150191103. PMID 9740045.
- ↑ „Proteomics: quantitative and physical mapping of cellular proteins“. Trends in Biotechnology. 17 (3): 121–127. март 1999. doi:10.1016/S0167-7799(98)01245-1. PMID 10189717.
- ↑ „Comprehensive Proteomic Analysis of Mesenchymal Stem Cell Exosomes Reveals Modulation of Angiogenesis via Nuclear Factor-KappaB Signaling“. Stem Cells. 34 (3): 601–613. март 2016. doi:10.1002/stem.2298. PMC 5785927. PMID 26782178.CS1-одржување: display-автори (link)
- ↑ „The Human Genome Project: big science transforms biology and medicine“. Genome Medicine (англиски). 5 (9): 79. 2013-09-13. doi:10.1186/gm483. PMC 4066586. PMID 24040834.
- ↑ „Protein analysis by shotgun/bottom-up proteomics“. Chemical Reviews. 113 (4): 2343–2394. април 2013. doi:10.1021/cr3003533. PMC 3751594. PMID 23438204.
- ↑ „Initial recommendations for performing, benchmarking and reporting single-cell proteomics experiments“. Nature Methods. 20 (3): 375–386. март 2023. doi:10.1038/s41592-023-01785-3. PMC 10130941 Проверете ја вредноста
|pmc=(help). PMID 36864200 Проверете ја вредноста|pmid=(help).CS1-одржување: display-автори (link) - ↑ „Single-cell protein analysis by mass spectrometry“. Current Opinion in Chemical Biology. Omics. 60: 1–9. февруари 2021. doi:10.1016/j.cbpa.2020.04.018. PMC 7767890 Проверете ја вредноста
|pmc=(help). PMID 32599342. - ↑ „Progress with gene-product mapping of the Mollicutes: Mycoplasma genitalium“. Electrophoresis. 16 (7): 1090–1094. јули 1995. doi:10.1002/elps.11501601185. PMID 7498152.CS1-одржување: display-автори (link)
- ↑ „Government backs proteome proposal“. Nature. 378 (6558): 653. декември 1995. Bibcode:1995Natur.378..653S. doi:10.1038/378653a0. PMID 7501000.
- ↑ „APAF - The Australian Proteome Analysis Facility“. www.proteome.org.au (англиски). Нов Јужен Велс, Австралија: Macquarie University. Архивирано од изворникот на 2017-02-06. Посетено на 2 февруари 2024.
- ↑ „Investigating the correspondence between transcriptomic and proteomic expression profiles using coupled cluster models“. Bioinformatics. 24 (24): 2894–2900. декември 2008. doi:10.1093/bioinformatics/btn553. PMC 4141638. PMID 18974169.
- ↑ „New frontiers in proteomics research: a perspective“. International Journal of Pharmaceutics. 299 (1–2): 1–18. август 2005. doi:10.1016/j.ijpharm.2005.04.010. PMID 15979831.
- ↑ „The major world of microRNAs“. мај 2003. Посетено на 2 февруари 2024.
- ↑ „Global, in vivo, and site-specific phosphorylation dynamics in signaling networks“. Cell. 127 (3): 635–648. ноември 2006. doi:10.1016/j.cell.2006.09.026. PMID 17081983.
- ↑ „Proteomics for cancer biomarker discovery“. Clinical Chemistry. 48 (8): 1160–1169. август 2002. PMID 12142368.
- ↑ „Correlation between protein and mRNA abundance in yeast“. Molecular and Cellular Biology. 19 (3): 1720–1730. март 1999. doi:10.1128/MCB.19.3.1720. PMC 83965. PMID 10022859.
- ↑ „Quantification of protein half-lives in the budding yeast proteome“. Proceedings of the National Academy of Sciences of the United States of America. 103 (35): 13004–13009. август 2006. Bibcode:2006PNAS..10313004B. doi:10.1073/pnas.0605420103. PMC 1550773. PMID 16916930.
- ↑ „Evaluation of multidimensional chromatography coupled with tandem mass spectrometry (LC/LC-MS/MS) for large-scale protein analysis: the yeast proteome“. Journal of Proteome Research. 2 (1): 43–50. 2003. CiteSeerX 10.1.1.460.237. doi:10.1021/pr025556v. PMID 12643542.
- ↑ „Large-scale analysis of the yeast proteome by multidimensional protein identification technology“. Nature Biotechnology. 19 (3): 242–247. март 2001. doi:10.1038/85686. PMID 11231557.
- ↑ „Repeatability and reproducibility in proteomic identifications by liquid chromatography-tandem mass spectrometry“. Journal of Proteome Research. 9 (2): 761–776. февруари 2010. doi:10.1021/pr9006365. PMC 2818771. PMID 19921851.CS1-одржување: display-автори (link)
- ↑ „Options and considerations when selecting a quantitative proteomics strategy“. Nature Biotechnology. 28 (7): 710–721. јули 2010. doi:10.1038/nbt.1661. PMID 20622845.
- ↑ „A Critical Review of Bottom-Up Proteomics: The Good, the Bad, and the Future of this Field“. Proteomes. 8 (3): 14. јули 2020. doi:10.3390/proteomes8030014. PMC 7564415 Проверете ја вредноста
|pmc=(help). PMID 32640657. - ↑ „Proteomics Is Analytical Chemistry: Fitness-for-Purpose in the Application of Top-Down and Bottom-Up Analyses“. Proteomes. 3 (4): 440–453. декември 2015. doi:10.3390/proteomes3040440. PMC 5217385. PMID 28248279.
- ↑ „Spectral quality overrides software score-A brief tutorial on the analysis of peptide fragmentation data for mass spectrometry laymen“. Journal of Mass Spectrometry. 56 (2): e4616. февруари 2021. doi:10.1002/jms.4616. PMID 32955142.
- ↑ „Good mass spectrometry and its place in good science“. Journal of Mass Spectrometry. 47 (6): 795–809. јуни 2012. Bibcode:2012JMSp...47..795D. doi:10.1002/jms.3038. PMID 22707172.
- ↑ „Selection and identification of single domain antibody fragments from camel heavy-chain antibodies“. FEBS Letters. 414 (3): 521–526. септември 1997. doi:10.1016/S0014-5793(97)01062-4. PMID 9323027.
- ↑ „DARPins: a new generation of protein therapeutics“. Drug Discovery Today. 13 (15–16): 695–701. август 2008. doi:10.1016/j.drudis.2008.04.013. PMID 18621567.
- ↑ „Aptamers: an emerging class of molecules that rival antibodies in diagnostics“. Clinical Chemistry. 45 (9): 1628–1650. септември 1999. doi:10.1093/clinchem/45.9.1628. PMID 10471678.
- ↑ „The Simoa HD-1 Analyzer: A Novel Fully Automated Digital Immunoassay Analyzer with Single-Molecule Sensitivity and Multiplexing“. Journal of Laboratory Automation. 21 (4): 533–547. август 2016. doi:10.1177/2211068215589580. PMID 26077162.CS1-одржување: display-автори (link)
- ↑ „Mass spectrometric immunoassay“. Analytical Chemistry. 67 (7): 1153–1158. април 1995. doi:10.1021/ac00103a003. OSTI 1175578. PMID 15134097.
- ↑ „SISCAPA, Stable Isotope Standard Capture with Anti-Peptide Antibodies“. Broad Institute of MIT and Harvard. 2015. Архивирано од изворникот на 2015-07-15. Посетено на 2 февруари 2024.
- ↑ 32,0 32,1 „Validation and development of fluorescence two-dimensional differential gel electrophoresis proteomics technology“. Proteomics. 1 (3): 377–396. март 2001. doi:10.1002/1615-9861(200103)1:3<377::AID-PROT377>3.0.CO;2-6. PMID 11680884.CS1-одржување: display-автори (link)
- ↑ „Insecticide-induced increase in the protein content of male accessory glands and its effect on the fecundity of females in the brown planthopper, Nilaparvata lugens Stål (Hemiptera: Delphacidae)“. Crop Protection. 29 (11): 1280–5. ноември 2010. doi:10.1016/j.cropro.2010.07.009.
- ↑ 34,0 34,1 „Proteomic analysis of insecticide triazophos-induced mating-responsive proteins of Nilaparvata lugens Stål (Hemiptera: Delphacidae)“. Journal of Proteome Research. 10 (10): 4597–4612. октомври 2011. doi:10.1021/pr200414g. PMID 21800909.
- ↑ 35,0 35,1 „Toward a definition of the complete proteome of plant peroxisomes: Where experimental proteomics must be complemented by bioinformatics“. Proteomics. 11 (9): 1764–1779. мај 2011. doi:10.1002/pmic.201000681. PMID 21472859.
- ↑ „Antibody-based proteomics for human tissue profiling“. Molecular & Cellular Proteomics. 4 (4): 384–393. април 2005. doi:10.1074/mcp.R500009-MCP200. PMID 15695805.
- ↑ „Modification-specific proteomics: characterization of post-translational modifications by mass spectrometry“. Current Opinion in Chemical Biology. 8 (1): 33–41. февруари 2004. doi:10.1016/j.cbpa.2003.12.009. PMID 15036154.
- ↑ 38,0 38,1 „Proteome of metastatic canine mammary carcinomas: similarities to and differences from human breast cancer“. Journal of Proteome Research. 9 (12): 6380–6391. декември 2010. doi:10.1021/pr100671c. PMID 20932060.
- ↑ „Proteomic analysis of differentially expressed protein in hemocytes of wild giant freshwater prawn Macrobrachium rosenbergii infected with infectious hypodermal and hematopoietic necrosis virus (IHHNV)“. Meta Gene. 5: 55–67. септември 2015. doi:10.1016/j.mgene.2015.05.004. PMC 4473098. PMID 26106581.
- ↑ 40,0 40,1 40,2 40,3 40,4 40,5 „Systems biology, proteomics, and the future of health care: toward predictive, preventative, and personalized medicine“. Journal of Proteome Research. 3 (2): 179–196. 2004. CiteSeerX 10.1.1.603.4384. doi:10.1021/pr0499693. PMID 15113093.
- ↑ Rozanova, Svitlana; Barkovits, Katalin; Nikolov, Miroslav; Schmidt, Carla; Urlaub, Henning; Marcus, Katrin (2021), Marcus, Katrin; Eisenacher, Martin; Sitek, Barbara (уред.), „Quantitative Mass Spectrometry-Based Proteomics: An Overview“, Quantitative Methods in Proteomics, Methods in Molecular Biology (англиски), New York, NY: Springer US, 2228: 85–116, doi:10.1007/978-1-0716-1024-4_8, ISBN 978-1-0716-1023-7, PMID 33950486 Проверете ја вредноста
|pmid=(help) - ↑ Nikolov, Miroslav; Schmidt, Carla; Urlaub, Henning (2012), Marcus, Katrin (уред.), „Quantitative Mass Spectrometry-Based Proteomics: An Overview“, Quantitative Methods in Proteomics, Methods in Molecular Biology (англиски), Totowa, NJ: Humana Press, 893: 85–100, doi:10.1007/978-1-61779-885-6_7, ISBN 978-1-61779-884-9, PMID 22665296, Посетено на 2 февруари 2024
- ↑ „Affinity proteomics: the role of specific binding reagents in human proteome analysis“. Expert Review of Proteomics. 9 (4): 401–414. август 2012. doi:10.1586/epr.12.34. PMID 22967077.
- ↑ „Selective enrichment of newly synthesized proteins for quantitative secretome analysis“. Nature Biotechnology. 30 (10): 984–990. октомври 2012. doi:10.1038/nbt.2356. PMID 23000932.
- ↑ „Bioorthogonal reactions for labeling proteins“. ACS Chemical Biology. 9 (1): 16–20. јануари 2014. doi:10.1021/cb4009292. PMID 24432752.
- ↑ „The Future of Bioorthogonal Chemistry“. ACS Central Science. 4 (8): 952–959. август 2018. doi:10.1021/acscentsci.8b00251. PMC 6107859. PMID 30159392.
- ↑ „Redefining clinical trials: the age of personalized medicine“. Cell. 148 (6): 1079–1080. март 2012. doi:10.1016/j.cell.2012.02.041. PMID 22424218.
- ↑ „Role of jasmonate in the rice (Oryza sativa L.) self-defense mechanism using proteome analysis“. Electrophoresis. 21 (12): 2492–2500. јули 2000. doi:10.1002/1522-2683(20000701)21:12<2492::AID-ELPS2492>3.0.CO;2-2. PMID 10939463.
- ↑ „New insights into plant responses to the attack from insect herbivores“. Annual Review of Genetics. 44: 1–24. 2010. doi:10.1146/annurev-genet-102209-163500. PMID 20649414.
- ↑ „Proteome Analysis of Rice (Oryza sativa L.) Mutants Reveals Differentially Induced Proteins during Brown Planthopper (Nilaparvata lugens) Infestation“. International Journal of Molecular Sciences. 14 (2): 3921–3945. февруари 2013. doi:10.3390/ijms14023921. PMC 3588078. PMID 23434671.CS1-одржување: display-автори (link)
- ↑ „How chemoproteomics can enable drug discovery and development“. Chemistry & Biology. 19 (1): 11–22. јануари 2012. doi:10.1016/j.chembiol.2012.01.001. PMC 3312051. PMID 22284350.
- ↑ de Mol NJ (2012). „Surface Plasmon Resonance for Proteomics“. Chemical Genomics and Proteomics. Methods in Molecular Biology. 800. стр. 33–53. doi:10.1007/978-1-61779-349-3_4. ISBN 978-1-61779-348-6. PMID 21964781.
- ↑ „Surface plasmon resonance mass spectrometry in proteomics“. Expert Review of Proteomics. 5 (3): 425–433. јуни 2008. doi:10.1586/14789450.5.3.425. PMID 18532910.
- ↑ 54,0 54,1 „Mass spectrometry-based proteomics and network biology“. Annual Review of Biochemistry. 81 (1): 379–405. 7 јули 2012. doi:10.1146/annurev-biochem-072909-100424. PMID 22439968.
- ↑ 55,0 55,1 „Mass spectrometry-based proteomics for systems biology“. Current Opinion in Biotechnology. 23 (4): 591–597. август 2012. doi:10.1016/j.copbio.2011.11.014. PMID 22169889.
- ↑ „What is Proteomics?“. ProteoConsult. Архивирано од изворникот на 2021-04-29. Посетено на 2024-02-02.Предлошка:MEDRSПристапено на 2 февруари 2024
- ↑ „What are biomarkers?“. Current Opinion in HIV and AIDS. 5 (6): 463–466. ноември 2010. doi:10.1097/COH.0b013e32833ed177. PMC 3078627. PMID 20978388.
- ↑ Biomarkers Definitions Working Group (март 2001). „Biomarkers and surrogate endpoints: preferred definitions and conceptual framework“. Clinical Pharmacology and Therapeutics. 69 (3): 89–95. doi:10.1067/mcp.2001.113989. PMID 11240971.
- ↑ „Proteomics in veterinary medicine: applications and trends in disease pathogenesis and diagnostics“. Veterinary Pathology. 51 (2): 351–362. март 2014. doi:10.1177/0300985813502819. PMID 24045891.
|hdl-access=бара|hdl=(help) - ↑ „Increased expression of BRCA2 and RAD51 in lymph node metastases of canine mammary adenocarcinomas“. Veterinary Pathology. 46 (3): 416–422. мај 2009. doi:10.1354/vp.08-VP-0212-K-FL. PMID 19176491.
- ↑ „Approaches to the study of the cell secretome“. Expert Review of Proteomics. 4 (2): 239–248. април 2007. doi:10.1586/14789450.4.2.239. PMID 17425459.
- ↑ „Whole proteome analysis of post-translational modifications: applications of mass-spectrometry for proteogenomic annotation“. Genome Research. 17 (9): 1362–1377. септември 2007. doi:10.1101/gr.6427907. PMC 1950905. PMID 17690205.CS1-одржување: display-автори (link)
- ↑ „Comparative proteogenomics: combining mass spectrometry and comparative genomics to analyze multiple genomes“. Genome Research. 18 (7): 1133–1142. јули 2008. doi:10.1101/gr.074344.107. PMC 2493402. PMID 18426904.CS1-одржување: display-автори (link)
- ↑ „UniProt“. www.uniprot.org.
- ↑ „ExPASy - PROSITE“. prosite.expasy.org.
- ↑ „A local average distance descriptor for flexible protein structure comparison“. BMC Bioinformatics. 15 (95): 95. април 2014. doi:10.1186/1471-2105-15-95. PMC 3992163. PMID 24694083.
- ↑ „A systematic framework for molecular dynamics simulations of protein post-translational modifications“. PLOS Computational Biology. 9 (7): e1003154. јули 2013. Bibcode:2013PLSCB...9E3154P. doi:10.1371/journal.pcbi.1003154. PMC 3715417. PMID 23874192.
- ↑ „Vienna-PTM web server: a toolkit for MD simulations of protein post-translational modifications“. Nucleic Acids Research. 41 (Web Server issue): W422–W426. јули 2013. doi:10.1093/nar/gkt416. PMC 3692090. PMID 23703210.
- ↑ „High-throughput discovery and characterization of fetal protein trafficking in the blood of pregnant women“. Proteomics. Clinical Applications. 3 (12): 1389–1396. декември 2009. doi:10.1002/prca.200900109. PMC 2825712. PMID 20186258.
- ↑ 70,0 70,1 Alterovitz G, Xiang M, Liu J, Chang A, Ramoni MF (2008). „System-wide peripheral biomarker discovery using information theory“. Biocomputing 2008. Pacific Symposium on Biocomputing. стр. 231–42. doi:10.1142/9789812776136_0024. ISBN 9789812776082. PMID 18229689.
- ↑ „Scaling Up Single-Cell Proteomics“. Molecular & Cellular Proteomics (English). 21 (1): 100179. јануари 2022. doi:10.1016/j.mcpro.2021.100179. PMC 8683604 Проверете ја вредноста
|pmc=(help). PMID 34808355 Проверете ја вредноста|pmid=(help).CS1-одржување: непрепознаен јазик (link) - ↑ „Increasing the throughput of sensitive proteomics by plexDIA“. Nature Biotechnology. 41 (1): 50–59. јули 2022. doi:10.1038/s41587-022-01389-w. PMC 9839897 Проверете ја вредноста
|pmc=(help). PMID 35835881 Проверете ја вредноста|pmid=(help).CS1-одржување: display-автори (link) - ↑ „Learning from natural variation across the proteomes of single cells“. PLOS Biology. 20 (1): e3001512. јануари 2022. doi:10.1371/journal.pbio.3001512. PMC 8765665 Проверете ја вредноста
|pmc=(help). PMID 34986167 Проверете ја вредноста|pmid=(help). - ↑ „Prioritized single-cell proteomics reveals molecular and functional polarization across primary macrophages“. bioRxiv: 2022.03.16.484655. 2022-03-18. doi:10.1101/2022.03.16.484655.CS1-одржување: display-автори (link)
- ↑ „Exploring functional protein covariation across single cells using nPOP“. Genome Biology. 23 (1): 261. декември 2022. doi:10.1186/s13059-022-02817-5. PMC 9756690 Проверете ја вредноста
|pmc=(help). PMID 36527135 Проверете ја вредноста|pmid=(help). - ↑ „TCPA: a resource for cancer functional proteomics data“. Nature Methods. 10 (11): 1046–1047. ноември 2013. doi:10.1038/nmeth.2650. PMC 4076789. PMID 24037243.CS1-одржување: display-автори (link)
- ↑ „The clinical plasma proteome: a survey of clinical assays for proteins in plasma and serum“. Clinical Chemistry. 56 (2): 177–185. февруари 2010. doi:10.1373/clinchem.2009.126706. PMID 19884488.
- ↑ „Six decades searching for meaning in the proteome“. Journal of Proteomics. 107: 24–30. јули 2014. doi:10.1016/j.jprot.2014.03.005. PMID 24642211.
- ↑ „Plasma Proteome Profiling to Assess Human Health and Disease“. Cell Systems. 2 (3): 185–195. март 2016. doi:10.1016/j.cels.2016.02.015. PMID 27135364.
|hdl-access=бара|hdl=(help) - ↑ „Large-scale inference of protein tissue origin in gram-positive sepsis plasma using quantitative targeted proteomics“. Nature Communications. 7: 10261. јануари 2016. Bibcode:2016NatCo...710261M. doi:10.1038/ncomms10261. PMC 4729823. PMID 26732734.
- ↑ „Proteomics reveals the effects of sustained weight loss on the human plasma proteome“. Molecular Systems Biology. 12 (12): 901. декември 2016. doi:10.15252/msb.20167357. PMC 5199119. PMID 28007936.CS1-одржување: display-автори (link)
- ↑ „Quantitative variability of 342 plasma proteins in a human twin population“. Molecular Systems Biology. 11 (1): 786. февруари 2015. doi:10.15252/msb.20145728. PMC 4358658. PMID 25652787.CS1-одржување: display-автори (link)
Библиографија[уреди | уреди извор]
- Arora PS, Yamagiwa H, Srivastava A, Bolander ME, Sarkar G (2005). „Comparative evaluation of two two-dimensional gel electrophoresis image analysis software applications using synovial fluids from patients with joint disease“. Journal of Orthopaedic Science. 10 (2): 160–166. doi:10.1007/s00776-004-0878-0. PMID 15815863. S2CID 45193214.
- Belhajjame K, Embury SM, Fan H, Goble CA, Hermjakob H, Hubbard SJ, и др. (September 2005). Proteome Data Integration: Characteristics and Challenges (PDF). Proceedings of the UK e-Science All Hands Meeting. Nottingham, UK. ISBN 978-1-904425-53-3. Архивирано од изворникот (PDF) на 28 June 2006.
- Ceciliani F, Eckersall D, Burchmore R, Lecchi C (March 2014). „Proteomics in veterinary medicine: applications and trends in disease pathogenesis and diagnostics“. Veterinary Pathology. 51 (2): 351–62. doi:10.1177/0300985813502819. hdl:2434/226049. PMID 24045891. S2CID 25693263.
- Liebler DC (2002). Introduction to proteomics: tools for the new biology. Totowa, NJ: Humana Press. ISBN 978-0-89603-992-6. ISBN 0-585-41879-9 (electronic, on Netlibrary?), ISBN 0-89603-991-9 hbk
- Macaulay IC, Carr P, Gusnanto A, Ouwehand WH, Fitzgerald D, Watkins NA (December 2005). „Platelet genomics and proteomics in human health and disease“. The Journal of Clinical Investigation. 115 (12): 3370–3377. doi:10.1172/JCI26885. PMC 1297260. PMID 16322782.
- Naven T, Westermeier R (2002). Proteomics in Practice: A Laboratory Manual of Proteome Analysis. Weinheim: Wiley-VCH. ISBN 978-3-527-30354-0. (focused on 2D-gels, good on detail)
- Twyman RM (2004). Principles Of Proteomics (Advanced Text Series). Oxford, UK: BIOS Scientific Publishers. ISBN 978-1-85996-273-2. (covers almost all branches of proteomics)
- von Hagen J (2008). Proteomics Sample Preparation. Weinheim: Wiley-VCH. ISBN 978-3-527-31796-7.
- Wilkins MR, Williams KL, Appel RD, Hochstrasser DF (1997). Proteome Research: New Frontiers in Functional Genomics (Principles and Practice). Berlin: Springer. ISBN 978-3-540-62753-1.
- Vasan RS (May 2006). „Biomarkers of cardiovascular disease: molecular basis and practical considerations“. Circulation. 113 (19): 2335–2362. doi:10.1161/CIRCULATIONAHA.104.482570. PMID 16702488.
Надворешни врски[уреди | уреди извор]
- Proteomics на Curlie (англиски)
