Хаплогрупа на ДНК на човечки Y-хромозом

Хаплогрупа на ДНК на човечки Y-хромозом — хаплогрупа дефинирана со мутации во не- рекомбинирачките делови на ДНК од машкиот специфичен Y хромозом (наречен Y-ДНК). Многу луѓе во хаплогрупата споделуваат сличен број на кратки тандем повторувања (STR) и типови на мутации наречени еднонуклеотидни полиморфизми (SNPs).[2]
Човечкиот Y-хромозом акумулира приближно две мутации по генерација.[3] Y-ДНК хаплогрупите претставуваат главни гранки на филогенетското дрво на Y-хромозомот кои споделуваат стотици или дури илјадници мутации уникатни за секоја хаплогрупа.
Најновиот заеднички предок на Y-хромозомот (Y-MRCA, неформално познат како Y-хромозомски Адам) е најновиот заеднички предок (MRCA) од кој сите моментално живи луѓе потекнуваат патрилинеално. Се проценува дека Y-хромозомскиот Адам живеел пред околу 236.000 години во Африка. Со испитување на другите тесни грла, повеќето евроазиски мажи (мажи од популации надвор од Африка) потекнуваат од човек кој живеел во Африка пред 69.000 години (Хаплогрупа КТ). Други големи тесни грла се случија пред околу 50.000 и 5.000 години и последователно потеклото на повеќето евроазиски мажи може да се проследи наназад до четири предци кои живееле пред 50.000 години, кои биле потомци на Африканците (E-M168).[4][5][6]
Конвенција за именување[уреди | уреди извор]
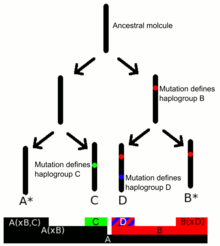
Y-ДНК хаплогрупите се дефинирани со присуство на серија Y-ДНК SNP маркери. Подкладите се дефинирани со терминален SNP, SNP најдалеку во филогенетското стебло на Y-хромозомот.[7][8] Конзорциумот на хромозомот Y (YCC) разви систем за именување на главните хаплогрупи на Y-ДНК со големи букви од А до Т, со понатамошни подклади именувани со помош на броеви и мали букви (номенклатура на долга рака YCC). Стенографската номенклатура на YCC ги именува Y-ДНК хаплогрупите и нивните подклади со првата буква од главната хаплогрупа Y-ДНК проследена со цртичка и името на дефинирачкиот терминал SNP.[9]
Номенклатурата на хаплогрупата на Y-ДНК се менува со текот на времето за да се приспособи на зголемениот број на SNP што се откриваат и тестираат, и како резултат на проширувањето на филогенетското дрво на Y-хромозомот. Оваа промена во номенклатурата резултираше со неконзистентна номенклатура која се користи во различни извори.[2] Оваа недоследност и сè понезгодната долга номенклатура, поттикна движење кон користење на поедноставната стенографија.
Филогенетска структура[уреди | уреди извор]
- Филогенетско дрво на Y-ДНК хаплогрупи[10]
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Главните хаплогрупи на Y-ДНК[уреди | уреди извор]
Хаплогрупите А и Б[уреди | уреди извор]
Хаплогрупата А е макрохаплогрупата NRY (некомбинирачка Y) од која потекнуваат сите модерни татковски хаплогрупи. Ретко е распространето во Африка, концентрирано меѓу популациите Коисани на југозапад и нилотските популации на североисток во долината на Нил. БТ е подклада на хаплогрупата А, поточно на кладата A1b (A2-T во Круциани и сор. 2011), и тоа:
- Хаплогрупа А
- Хаплогрупа А00
- Хаплогрупа А0 (порано и А1б)
- Хаплогрупа А1 (исто така А1а-Т)
- Хаплогрупа А1а (M31)
- Хаплогрупа A1b (исто така A2-T; P108, V221)
- Хаплогрупа A1b1a1 (исто така A2; M14)
- Хаплогрупа A1b1b (исто така A3; M32)
- Хаплогрупа БТ (M91, M42, M94, M139, M299)
- Хаплогрупа Б (M60)
- Хаплогрупа КТ
Хаплогрупа КТ (P143)[уреди | уреди извор]
Дефинитивните мутации што го одвојуваат КТ (сите хаплогрупи освен А и Б) се M168 и M294. Местото на потекло е веројатно во Африка. Неговата старост се проценува на приближно 88.000 години,[11][12] а во поново време на околу 100.000[13] или 101.000 години.[14]
Хаплогрупа C (M130)[уреди | уреди извор]
- Хаплогрупа C (M130, M216) пронајдена во Азија, Океанија и Северна Америка
- Хаплогрупа C1 (F3393/Z1426)
- Хаплогрупа C1a (CTS11043)
- Хаплогрупа C1b (F1370, Z16480)
- Хаплогрупа C1b1 (AM00694/K281)
- Хаплогрупа C1b1a (B66/Z16458)
- Хаплогрупа C1b1a1 (M356) пронајдена со ниска фреквенција во Јужна Азија, Југозападна Азија и северна Кина
- Хаплогрупа C1b1a2 (B65)
- Хаплогрупата C1b1a2a (B67) пронајдена меѓу луѓето Лебо во Борнео, Индонезија
- Хаплогрупа C1b1a2b (F725) пронајдена кај Хан Кинезите (Гуангдонг, Хунан и Шанкси), луѓето Даи (Јунан), луѓето Мурут (Брунеи), Малајците (Сингапур) и луѓето Аета (Филипини)
- Хаплогрупа C1b1a3 (Z16582) пронајдена со ниска фреквенција во Саудиска Арабија и Ирак
- Хаплогрупата C1b1b (B68) пронајдена кај луѓето од Дусун (Брунеи)
- Хаплогрупа C1b1a (B66/Z16458)
- Хаплогрупа C1b2 (C-Z16582)
- Хаплогрупа C1b3 (B477/Z31885)
- Хаплогрупа C1b3a (M38) пронајдена во Индонезија, Нова Гвинеја, Меланезија, Микронезија и Полинезија
- Хаплогрупа C1b3b (M347, P309) Пронајдена кај домородните народи во Австралија
- Хаплогрупа C1b1 (AM00694/K281)
- Хаплогрупа C2 (M217, P44) Пронајдена низ Евроазија и Северна Америка, но особено кај Монголите, Казахстанците, Тунгуските народи, Палеосиберите и народите кои зборуваат На-Дене
- Хаплогрупа C1 (F3393/Z1426)
Хаплогрупа Д (CTS3946)[уреди | уреди извор]
- Хаплогрупа Д (CTS3946)
- Хаплогрупа D1 (M174) пронајдена во Јапонија, Кина (особено Тибет), Андаманските Острови
- Хаплогрупа D1a (CTS11577)
- Хаплогрупа D1a1 (Z27276, Z27283, Z29263)
- Хаплогрупа D1a1a (M15) Се наоѓа главно кај Тибетанците, народите Кианги, Ји и Хмонг-Миен
- Хаплогрупа D1a1b (P99) Пронајдена главно кај Тибетанците, Киангските народи, Наксите и Турците
- Хаплогрупа D1a2 (M55, M57, M64.1, M179, P12, P37.1, P41.1 (M359.1), 12f2.2) Се наоѓа главно во Јапонија
- Хаплогрупа D1a3 (Y34637) пронајдена кај андаманските народи (Онге, Јарава)
- Хаплогрупа D1a1 (Z27276, Z27283, Z29263)
- Хаплогрупа D1b (L1366, L1378, M226.2) пронајдена на островот Мактан, Филипини
- Хаплогрупа D1a (CTS11577)
- Хаплогрупа D2 (A5580.2) пронајдена во Нигерија, Саудиска Арабија и Сирија
- Хаплогрупа D1 (M174) пронајдена во Јапонија, Кина (особено Тибет), Андаманските Острови
Хаплогрупа Е (M96)[уреди | уреди извор]
- Хаплогрупа Е (М40, М96) Пронајдена во Африка и делови од Блискиот Исток и Европа
- Хаплогрупа Е1 (P147)
- Хаплогрупа Е1а (М33, М132) порано Е1
- Хаплогрупа E1b (P177)
- Хаплогрупа E1b1 (P2, DYS391p); порано Е3
- Хаплогрупа E1b1a (V38)
- Хаплогрупа E1b1a1 (M2) Пронајдена во Африка, особено меѓу популациите што зборуваат Нигер-Конго.; порано E3a
- Хаплогрупа E1b1a2 (M329) Пронајдена во Африка, особено во Етиопија кај популациите што зборуваат омотски.; порано E3*
- Хаплогрупа E1b1b (M215)
- Хаплогрупа E1b1b1 (M35) Пронајдена во Рогот на Африка, Северна Африка, Блискиот Исток и Европа (особено во областите во близина на Медитеранот и на Балканот); порано E3b
- Хаплогрупа E1b1a (V38)
- Хаплогрупа E1b1 (P2, DYS391p); порано Е3
- Хаплогрупа Е2 (M75)
- Хаплогрупа Е1 (P147)
Хаплогрупа F (M89)[уреди | уреди извор]
Групите кои потекнуваат од хаплогрупата F се наоѓаат кај околу 90% од светската популација, но речиси исклучиво надвор од субсахарска Африка.
Ф xG, H, I, J, K е ретка кај модерните популации и врвови во Јужна Азија, особено Шри Ланка.[10] Исто така, се чини дека одамна е присутна во Југоисточна Азија; пријавен е со стапки од 4-5% во Сулавеси и Лембата. Една студија, која не проверуваше сеопфатно за други подклади на F-M89 (вклучувајќи некои подклади на GHIJK), покажа дека индонезиските мажи со SNP P14/PF2704 (што е еквивалентно на M89), сочинуваат 1,8% од мажите во Западен Тимор, 1,5% од Флорес 5,4% од Лембата 2,3% од Сулавеси и 0,2% во Суматра.[15][16] F* (Ф xF1, F2, F3) е забележано кај 10% од мажите во Шри Ланка и Јужна Индија, 5% во Пакистан, како и пониски нивоа кај народот Таманг (Непал) и во Иран. F1 (P91), F2 (M427) и F3 (M481; претходно F5) сите се многу ретки и практично ексклузивни за регионите/етничките малцинства во Шри Ланка, Индија, Непал, Јужна Кина, Тајланд, Бурма и Виетнам. Меѓутоа, во такви случаи, можноста за погрешна идентификација се смета за релативно висока и некои може да припаѓаат на погрешно идентификувани подклади на Хаплогрупата GHIJK.[17]
Хаплогрупа G (M201)[уреди | уреди извор]
Хаплогрупата G (M201) настанала пред околу 48.000 години, а нејзиниот најнов заеднички предок најверојатно живеел пред 26.000 години на Блискиот Исток. Во Европа се прошири со неолитската револуција.
Го има во многу етнички групи во Евроазија; најчесто на Кавказ, Иран, Анадолија и Левант. Се среќава во скоро сите европски земји, но најчесто во Гагаузија, југоисточна Романија, Грција, Италија, Шпанија, Португалија, Тирол и Бохемија со највисоки концентрации на некои медитерански острови; невообичаено во Северна Европа.[18][19]
G-M201 се наоѓа во мал број и во северозападна Кина и Индија, Бангладеш, Пакистан, Шри Ланка, Малезија и Северна Африка.
- Хаплогрупа Г1
- Хаплогрупа Г2
- Хаплогрупа G2a
- Хаплогрупа G2a1
- Хаплогрупа G2a2
- Хаплогрупа G2a3
- Хаплогрупа G2a3a
- Хаплогрупа G2a3b
- Хаплогрупа G2b
- Хаплогрупа G2c (порано Хаплогрупа G5)
- Хаплогрупа G2c1
- Хаплогрупа G2a
Хаплогрупа H (M69)[уреди | уреди извор]
Хаплогрупата H (M69) веројатно се појавила во Јужна Централна Азија, Јужна Азија или Западна Азија, околу 48.000 години БП, и останува во голема мера распространета таму во формите на H1 (M69) и H3 (Z5857). Нејзините под-клади се наоѓаат и на пониски фреквенции во Иран, Централна Азија, низ Блискиот Исток и Арапскиот полуостров.
Сепак, H2 (P96) е присутен во Европа од неолитот и H1a1 (M82) се шират на запад во средновековната ера со миграцијата на Ромите.
Хаплогрупа I (M170)[уреди | уреди извор]
Хаплогрупата I (M170, M258) се наоѓа главно во Европа и Кавказ.
- Хаплогрупа I1 нордиски/нордиски европиди (M253) Пронајден главно во северна Европа
- Хаплогрупа I2 Динариди/Динариски европиди (Р215) Се среќава главно на Балканот, југоисточна Европа и Сардинија освен за I2B1 (m223) кој се среќава со умерена фреквенција во Западна, Централна и Северна Европа.
Хаплогрупа Ј (M304)[уреди | уреди извор]
Хаплогрупата Ј (M304, S6, S34, S35) се наоѓа главно на Блискиот Исток, Кавказ и Југоисточна Европа.
- Хаплогрупата Ј* (J-M304*) е ретка надвор од островот Сокотра.
- Хаплогрупата Ј1 Семити/Бедуини Арабиди (М267) се поврзани со североисточните кавкаски народи во Дагестан и семитските јазици кои зборуваат луѓе на Блискиот Исток, Етиопија и Северна Африка, а исто така се наоѓаат во Медитеранска Европа на помали фреквенции слично како хаплогрупата Т.
- Хаплогрупата Ј2 Сириди/Нахреидни Арабиди (М172) се среќава главно кај народите кои зборуваат семитски, Анадолија, Грција, Балканот, Италија, Иран, Кавказ, Јужна Азија и Централна Азија.
Хаплогрупа K (M9)[уреди | уреди извор]
Хаплогрупата К (М9) е распространета низ цела Евроазија, Океанија и меѓу Индијанците.
K(xLT, K2a, K2b) - односно K*, K2c, K2d или K2e - се наоѓа главно во Меланезија, Абориџините Австралијци, Индија, Полинезија и островот Југоисточна Азија.
Хаплогрупи L и T (K1)[уреди | уреди извор]
Хаплогрупата L (M20) се наоѓа во Јужна Азија, Централна Азија, Југозападна Азија и Медитеранот.
Хаплогрупата Т (M184, M70, M193, M272) се наоѓа на високи нивоа во Африканскиот рог (главно народи кои зборуваат кушитски), делови од Јужна Азија, Блискиот Исток и Медитеранот. T-M184 се наоѓа и кај значајните малцинства на Сциаценси, Стилфсер, Египќани, Оманци, Сефардските Евреи,[20] Ибијци и Тоубоу. На ниски фреквенции го има и во други делови на Медитеранот и Јужна Азија.
Хаплогрупа K2 (K-M526)[уреди | уреди извор]
Единствените живи мажјаци пријавени дека ја носат основната парагрупа К2* се домородните Австралијци. Големите студии објавени во 2014 и 2015 година сугерираат дека до 27% од Абориџините мажи од Австралија носат К2*, додека други носат подклада на К2.
Хаплогрупи K2a, K2a1, NO и NO1[уреди | уреди извор]
Хаплогрупата Н[уреди | уреди извор]
Хаплогрупата N (M231) се наоѓа во северна Евроазија, особено кај говорителите на уралските јазици.
Хаплогрупата N веројатно потекнува од источна Азија и се проширила и на север и на запад во Сибир, што е најчестата група пронајдена кај некои народи што зборуваат уралски.
Хаплогрупата О[уреди | уреди извор]
Хаплогрупата О (М175) се среќава со најголема фреквенција во Источна Азија и Југоисточна Азија, со пониски фреквенции во Јужен Пацифик, Централна Азија, Јужна Азија и острови во Индискиот Океан (на пр. Мадагаскар, Коморите).
- Хаплогрупа О1 (F265/M1354, CTS2866, F75/M1297, F429/M1415, F465/M1422)
- Хаплогрупа O1a (M119, CTS31, F589/Page20, L246, L466) Пронајдена во источна, централна и јужна континентална Кина, Тајван и Југоисточна Азија, особено кај австронезиските и таи-кадаи народи
- Хаплогрупа O1b (P31, M268)
- Хаплогрупа O1b1 (M95) пронајдена во Јапонија, Кина, Тајван, Југоисточна Азија и Индискиот потконтинент, особено кај народите што зборуваат австроазиски и таи-кадаи, Малејците и Индонезијците
- Хаплогрупа O1b2 (SRY465, M176) пронајдена во Јапонија, Кореја, Манџурија и Југоисточна Азија
- Хаплогрупа О2 (М122) пронајдена низ Источна Азија, Југоисточна Азија и Австронезија вклучувајќи ја Полинезија
Хаплогрупи K2b1, M & S[уреди | уреди извор]
Не се идентификувани примери на базалната парагрупа K2b1*. Мажјаците кои носат потклади на K2b1 се наоѓаат првенствено кај папуанските народи, микронезиските народи, домородните Австралијци и Полинезијците.
Нејзините примарни подклади се две главни хаплогрупи:
- Хаплогрупа S (B254) позната и како K2b1a: пронајдена во висорамнините на Папуа Нова Гвинеја и;
- Хаплогрупа М (P256) позната и како K2b1b: пронајдена во Нова Гвинеја и Меланезија.
Хаплогрупа P (K2b2)[уреди | уреди извор]
Хаплогрупата P (P295) има две примарни гранки: P1 (P-M45) и исклучително ретката P2 (P-B253).[21]
P*, P1* и P2 се наоѓаат заедно само на островот Лузон на Филипините.[21] Особено, P* и P1* се наоѓаат со значителни стапки меѓу членовите на народот Аета (или Агта) од Лузон.[22] Додека, P1* сега е почеста кај живите индивидуи во Источен Сибир и Централна Азија, исто така се наоѓа на ниски нивоа во копното Југоисточна Азија и Јужна Азија. Разгледани заедно, овие дистрибуции имаат тенденција да сугерираат дека P* се појавил од K2b во Југоисточна Азија.[22][23]
P1 е исто така главен јазол на два примарни клада:
- Хаплогрупа Q (Q-M242) и;
- Хаплогрупа R (R-M207). Тие го делат заедничкиот маркер M45 како додаток на најмалку 18 други SNP.
Хаплогрупа Q (MEH2, M242, P36) пронајдена во Сибир и Америка Хаплогрупа R (M207, M306): пронајдена во Европа, Западна Азија, Централна Азија и Јужна Азија.
Хаплогрупа Q M242[уреди | уреди извор]
Q е дефиниран од SNP M242. Се верува дека се појавил во Централна Азија пред околу 32.000 години.[24][25] Подкладите на Хаплогрупата Q со нивните дефинирани мутации, според ISOGG дрвото од 2008 година[26] се дадени подолу. ss4 bp, rs41352448, не е претставена во дрвото ISOGG 2008 бидејќи е вредност за STR. Оваа вредност на ниска фреквенција е пронајдена како нова лоза Q (Q5) кај индиските популации.[27]
Дрвото ISOGG од 2008 година
- Q (M242)
- П*
- Q1 (P36.2)
- Q1*
- Q1a (MEH2)
- Q1a*
- Q1a1 (M120, M265/N14) Пронајден со мала фреквенција кај Бутанците,[28] Дунгани, Хан Кинезите, Јапонците, Корејците, Монголците,[29] Накси и Тибетанците[30][31]
- Q1a2 (M25, M143) Пронајден на мала до умерена фреквенција кај некои популации од Југозападна Азија, Централна Азија и Сибир
- Q1a3 (M346)
- Q1a3* Пронајден на ниска фреквенција во Пакистан, Индија и Тибет
- Q1a3a (M3) Типично за домородните народи на американскиот континент
- Q1a3a*
- Q1a3a1 (M19) Пронајден кај некои домородни народи во Јужна Америка, како што се Тикуна и Вајуу[32]
- Q1a3a2 (M194)
- Q1a3a3 (M199, P106, P292)
- Q1a4 (P48)
- Q1a5 (P89)
- Q1a6 (M323) Пронајден во значително малцинство јеменски Евреи
- Q1b (M378) Пронајден на ниска фреквенција меѓу примероците на Хазара и Синди
Хаплогрупа R (M207)[уреди | уреди извор]

Хаплогрупата R е дефинирана со SNP M207. Најголемиот дел од Хаплогрупата R е претставена во потомокот R1 (M173), кој потекнува од Сибир. R1 има две потклади: R1a и R1b.
R1a е поврзан со прото-индо-иранските и балто-словенските народи и сега се наоѓа првенствено во Средна Азија, Јужна Азија и Источна Европа.
Хаплогрупата R1b е доминантна хаплогрупа во Западна Европа и исто така се среќава ретко распространета меѓу различни народи од Азија и Африка. Нејзината потклада R1b1a2 (M269) е хаплогрупата која најчесто се среќава кај современите западноевропски популации и е поврзана со итало-келтските и германските народи.
- Хаплогрупа R1 (M173) Пронајдена низ западна Евроазија
- Хаплогрупа R1a (M420) пронајдена во Централна Азија, Јужна Азија и Централна, Северна и Источна Европа, Балканот
- Хаплогрупа R1b (M343) пронајдена во Западна Европа, Западна Азија, Централна Азија, Северна Африка и северен Камерун
- Хаплогрупата R2 (M124) пронајдена во Јужна Азија, Кавказ, Централна Азија и Источна Европа
Хронолошки развој на хаплогрупите[уреди | уреди извор]
| Хаплогрупа | Можно време на потекло | Можно место на потекло | Можно ВНЗП[33][12] |
|---|---|---|---|
| A00 | 235,900[5] или 275,000 п.н.е.[34] | Африка[35] | 235,900 п.н.е. |
| BT | 130,700 п.н.е.[5] | Африка | 88,000 п.н.е. |
| CT | 88,000[5] или 101-100,000 п.н.е.[13][14] | Африка | 68,500 п.н.е. |
| E | 65,200,[5] 69,000,[36] или 73,000 п.н.е.[37] | Источна Африка[38] или Азија[15] | 53,100 п.н.е. |
| F | 65,900 п.н.е.[5] | Евроазија | 48,800 п.н.е. |
| G | 48,500 п.н.е.[5] | Среден Исток | 26,200 п.н.е. |
| IJ | 47,200 п.н.е.[5] | Среден Исток | 42,900 п.н.е. |
| K | 47,200 п.н.е.[5] | Азија | 45,400 п.н.е. |
| P | 45,400 п.н.е.[5] | Азија | 31,900 п.н.е. |
| J | 42,900 п.н.е.[5][39] | Среден Исток | 31,600 п.н.е. |
| I | 42,900 п.н.е.[5] | Европа | 27,500 п.н.е. |
| E-M215 (E1b1b) | 42,300 п.н.е.[5][40] | Источна Африка | 34,800 п.н.е. |
| E-V38 (E1b1a) | 42,300 п.н.е.[5][40] | Источна Африка | 40,100 п.н.е. |
| N | 36,800 п.н.е.[5][41] | Азија | 22,100 п.н.е. |
| E1b1b-M35 | 34,800 п.н.е.[5][40] | Источна Африка | 24,100 п.н.е. |
| R | 31,900 п.н.е.[5] | Азија | 28,200 п.н.е. |
| J-M267 (J1) | 31,600 п.н.е.[5][39] | Среден Исток | 18,500 п.н.е. |
| J-M172 (J2) | 31,600 п.н.е.[5][39] | Среден Исток | 27,800 п.н.е.[5][42] |
| R-M173 (R1) | 28,200 п.н.е.[5] | Азија | 22,800 п.н.е. |
| I-M253 (I1) | 27,500 п.н.е.[5][43][44] | Европа | 4,600 п.н.е. |
| I-M438 (I2) | 27,500 п.н.е.[5] | Европа | 21,800 п.н.е. |
| R-M420 (R1a) | 22,800 п.н.е.[5][45] | Евроазија | 18,300 п.н.е. |
| R-M343 (R1b) | 22,800 п.н.е.[5] | Евроазија | 20,400 п.н.е. |
| I2-L460 (I2a) | 21,800 п.н.е.[5][46] | Европа | 21,100 п.н.е. |
| I2a-P37 | 21,100 п.н.е.[5][43][47] | Европа | 18,500 п.н.е. |
| E1b1b-M78 | 19,800 п.н.е.[5][40][48] | Северна Африка[48] | 13,400 п.н.е.[5][48] |
| I2a-M423 | 18,500 п.н.е.[5][47] | Европа | 13,500 п.н.е. |
| I2a-M223 | 17,400 п.н.е.[5] | Европа | 12,100 п.н.е. |
| N1c-M178 | 14,200 п.н.е.[5][41] | Азија | 11,900 п.н.е. |
| R1a-M17 | 14,100 п.н.е.[5][45][49] | Источна Европа | 8,500 п.н.е. |
| R1b-M269 | 13,300 п.н.е.[5] | Источна Европа | 6,400 п.н.е.[50] |
| E1b1b-V12 | 11,800 п.н.е.[5][48] | Северна Африка | 9,900 п.н.е. |
| E-U175 (E1b1a8) | 9,200 п.н.е.[5][40] | Источна Африка | 8,500 п.н.е. |
| E1b1b-V13 | 8,100 п.н.е.[5] | Јужна Европа | 4,800 п.н.е. |
| E-M191 (E1b1a7) | 7,400 п.н.е.[5][40] | Источна Африка | 6,400 п.н.е. |
| E-U174 (E1b1a-U174) | 6,400 п.н.е.[5][40] | Источна Африка | 5,300 п.н.е. |
| R1b-L151 | 5,800 п.н.е.[5] | Источна Европа | 4,800 п.н.е. |
| R1a-Z280 | 5,000 п.н.е.[5] | Источна Европа | 4,600 п.н.е.[51] |
| R1a-M458 | 4,700 п.н.е.[5] | Источна Европа | 4,700 п.н.е.[51] |
Наводи[уреди | уреди извор]
- ↑ Hallast P, Agdzhoyan A, Balanovsky O, Xue Y, Tyler-Smith C (2020). „A Southeast Asian origin for present-day non-African human Y chromosomes“. Hum Genet. 140 (2): 299–307. doi:10.1007/s00439-020-02204-9. PMC 7864842 Проверете ја вредноста
|pmc=(help). PMID 32666166.CS1-одржување: повеќе имиња: список на автори (link) - ↑ 2,0 2,1 „Understanding Haplogroups: How are the haplogroups named?“. Family Tree DNA. Архивирано од изворникот на 21 June 2012. Посетено на 31 March 2013.
- ↑ Dolgin, Elie (2009). „Human mutation rate revealed“. Nature. doi:10.1038/news.2009.864. Посетено на 18 September 2017. "one mutation in every 30 million base pairs"
- ↑ Karmin; и др. (2015). „A recent bottleneck of Y chromosome diversity coincides with a global change in culture“. Genome Research. 25 (4): 459–66. doi:10.1101/gr.186684.114. PMC 4381518. PMID 25770088. "we date the Y-chromosomal most recent common ancestor (MRCA) in Africa at 254 (95% CI 192–307) kya and detect a cluster of major non-African founder haplogroups in a narrow time interval at 47–52 kya, consistent with a rapid initial colonization model of Eurasia and Oceania after the out-of-Africa bottleneck. In contrast to demographic reconstructions based on mtDNA, we infer a second strong bottleneck in Y-chromosome lineages dating to the last 10 ky. We hypothesize that this bottleneck is caused by cultural changes affecting variance of reproductive success among males."
- ↑ 5,00 5,01 5,02 5,03 5,04 5,05 5,06 5,07 5,08 5,09 5,10 5,11 5,12 5,13 5,14 5,15 5,16 5,17 5,18 5,19 5,20 5,21 5,22 5,23 5,24 5,25 5,26 5,27 5,28 5,29 5,30 5,31 5,32 5,33 5,34 5,35 5,36 5,37 5,38 5,39 5,40 5,41 „YFull YTree“. YFull. Посетено на 15 September 2017.
- ↑ „Something Weird Happened to Men 7,000 Years Ago, And We Finally Know Why“. 31 May 2018.
Around 7000 years ago - all the way back in the Neolithic - something really peculiar happened to human genetic diversity. Over the next 2,000 years, and seen across Africa, Europe and Asia, the genetic diversity of the Y chromosome collapsed, becoming as though there was only one man for every 17 women.
- ↑ „Understanding Results: Y-DNA Single Nucleotide Polymorphism (SNP): What is a Y-chromosome DNA (Y-DNA) haplogroup?“. Family Tree DNA. Посетено на 31 March 2013.
Y-chromosome DNA (Y-DNA) haplogroups are the major branches on the human paternal family tree. Each haplogroup has many subbranches. These are subclades.
- ↑ „myFTDNA 2.0 User Guide: Y-DNA: What is the Y-DNA – Matches page?“. Family Tree DNA. Посетено на 31 March 2013.
A terminal SNP determines the terminal (final) subbranch on the Y-DNA Tree to which someone belongs.
- ↑ „Understanding Results: Y-DNA Single Nucleotide Polymorphism (SNP): How are haplogroups and their subclades named?“. Family Tree DNA. Посетено на 31 March 2013.
- ↑ 10,0 10,1 Copyright 2015 ISOGG. „ISOGG 2015 Y-DNA Haplogroup Tree Trunk“. isogg.org.
- ↑ Underhill and Kivisild; Kivisild, T (2007). „Use of Y Chromosome and Mitochondrial DNA Population Structure in Tracing Human Migrations“. Annu. Rev. Genet. 41 (1): 539–64. doi:10.1146/annurev.genet.41.110306.130407. PMID 18076332.
- ↑ 12,0 12,1 Karafet, TM; Mendez, FL; Meilerman, MB; Underhill, PA; Zegura, SL; Hammer, MF (2008). „New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree“. Genome Research. 18 (5): 830–38. doi:10.1101/gr.7172008. PMC 2336805. PMID 18385274.
- ↑ 13,0 13,1 „A recent bottleneck of Y chromosome diversity coincides with a global change in culture“. Genome Research. 25 (4): 459–66. April 2015. doi:10.1101/gr.186684.114. PMC 4381518. PMID 25770088.
- ↑ 14,0 14,1 „A Rare Deep-Rooting D0 African Y-chromosomal Haplogroup and its Implications for the Expansion of Modern Humans Out of Africa“. Genetics. 212 (4): 1421–28. June 2019. doi:10.1534/genetics.119.302368. PMC 6707464. PMID 31196864.
- ↑ 15,0 15,1 Chiaroni, Jacques; Underhill, Peter A.; Cavalli-Sforza, Luca L. (1 December 2009). „Y chromosome diversity, human expansion, drift, and cultural evolution“. Proceedings of the National Academy of Sciences of the United States of America. 106 (48): 20174–79. Bibcode:2009PNAS..10620174C. doi:10.1073/pnas.0910803106. PMC 2787129. PMID 19920170.
- ↑ Tumonggor, Meryanne K (2014). „Isolation, contact and social behavior shaped genetic diversity in West Timor“. Journal of Human Genetics. 59 (9): 494–503. doi:10.1038/jhg.2014.62. PMC 4521296. PMID 25078354.
- ↑ This was, for instance, the case with the original subclade F3 (M96), which has since been renamed Haplogroup H2.
- ↑ Passarino, Giuseppe; Cavalleri, Gianpiero L; Lin, Alice A; Cavalli-Sforza, LL; Børresen-Dale, AL; Underhill, PA (2002). „Different genetic components in the Norwegian population revealed by the analysis of mtDNA and Y chromosome polymorphisms“. European Journal of Human Genetics. 10 (9): 521–29. doi:10.1038/sj.ejhg.5200834. PMID 12173029.
- ↑ Karlsson, Andreas O; Wallerström, Thomas; Götherström, Anders; Holmlund, Gunilla (2006). „Y-chromosome diversity in Sweden – A long-time perspective“. European Journal of Human Genetics. 14 (8): 963–70. doi:10.1038/sj.ejhg.5201651. PMID 16724001.
- ↑ Nogueiro, Inês (2009). „Phylogeographic analysis of paternal lineages in NE Portuguese Jewish communities“. American Journal of Physical Anthropology. 141 (3): 373–81. doi:10.1002/ajpa.21154. PMID 19918998.
- ↑ 21,0 21,1 ISOGG, 2016, Y-DNA Haplogroup P and its Subclades – 2016 (20 June 2016).
- ↑ 22,0 22,1 Tumonggor, Meryanne K; Karafet, Tatiana M; Downey, Sean; Lansing, J Stephen; Norquest, Peter; Sudoyo, Herawati; Hammer, Michael F; Cox, Murray P (31 July 2014). „Isolation, contact and social behavior shaped genetic diversity in West Timor“. Journal of Human Genetics. 59 (9): 494–503. doi:10.1038/jhg.2014.62. PMC 4521296. PMID 25078354.
- ↑ Tatiana M Karafet; и др. (2015). „Improved phylogenetic resolution and rapid diversification of Y-chromosome haplogroup K-M526 in Southeast Asia“. European Journal of Human Genetics. 23 (3): 369–73. doi:10.1038/ejhg.2014.106. PMC 4326703. PMID 24896152.
- ↑ Fagundes, Nelson J.R.; Ricardo Kanitz; Roberta Eckert; Ana C.S. Valls; Mauricio R. Bogo; Francisco M. Salzano; David Glenn Smith; Wilson A. Silva; Marco A. Zago (2008). „Mitochondrial Population Genomics Supports a Single Pre-Clovis Origin with a Coastal Route for the Peopling of the Americas“ (PDF). American Journal of Human Genetics. 82 (3): 583–92. doi:10.1016/j.ajhg.2007.11.013. PMC 2427228. PMID 18313026. Архивирано од изворникот (PDF) на 2009-03-25. Посетено на 2013-05-22.
Since the first studies, it has been found that extant Native American populations exhibit almost exclusively five "mtDNA haplogroups" (A–D and X)6 classified in the autochthonous haplogroups A2, B2, C1, D1, and X2a.7 Haplogroups A–D are found all over the New World and are frequent in Asia, supporting a northeastern Asian origin of these lineages
- ↑ Zegura, S. L.; Karafet, TM; Zhivotovsky, LA; Hammer, MF (2003). „High-Resolution SNPs and Microsatellite Haplotypes Point to a Single, Recent Entry of Native American Y Chromosomes into the Americas“. Molecular Biology and Evolution. 21 (1): 164–75. doi:10.1093/molbev/msh009. PMID 14595095.
- ↑ „Y-DNA Haplogroup Tree 2010“. International Society of Genetic Genealogy. Посетено на 1 July 2010.
- ↑ Sharma, Swarkar; Rai, Ekta; Bhat, Audesh K; Bhanwer, Amarjit S; Bamezaicorresponding, Rameshwar NK (2007). „A novel subgroup Q5 of human Y-chromosomal haplogroup Q in India“. BMC Evol Biol. 7: 232. doi:10.1186/1471-2148-7-232. PMC 2258157. PMID 18021436.
- ↑ Hallast, P.; Batini, C.; Zadik, D.; Maisano Delser, P.; Wetton, J. H.; Arroyo-Pardo, E.; Cavalleri, G. L.; De Knijff, P.; Destro Bisol, G. (2015). „The Y-Chromosome Tree Bursts into Leaf: 13,000 High-Confidence SNPs Covering the Majority of Known Clades“. Molecular Biology and Evolution. 32 (3): 661–73. doi:10.1093/molbev/msu327. PMC 4327154. PMID 25468874.
- ↑ Di Cristofaro, Julie; Pennarun, Erwan; Mazières, Stéphane; Myres, Natalie M.; Lin, Alice A.; Temori, Shah Aga; Metspalu, Mait; Metspalu, Ene; Witzel, Michael (2013). „Afghan Hindu Kush: Where Eurasian Sub-Continent Gene Flows Converge“. PLOS ONE. 8 (10): e76748. Bibcode:2013PLoSO...876748D. doi:10.1371/journal.pone.0076748. PMC 3799995. PMID 24204668.
- ↑ „Genetic evidence supports demic diffusion of Han culture“. Nature. 431 (7006): 302–05. September 2004. Bibcode:2004Natur.431..302W. doi:10.1038/nature02878. PMID 15372031.
- ↑ „The Eurasian heartland: a continental perspective on Y-chromosome diversity“. Proc. Natl. Acad. Sci. U.S.A. 98 (18): 10244–49. August 2001. Bibcode:2001PNAS...9810244W. doi:10.1073/pnas.171305098. PMC 56946. PMID 11526236.
- ↑ „Y-chromosome evidence for differing ancient demographic histories in the Americas“. Am. J. Hum. Genet. 73 (3): 524–39. September 2003. doi:10.1086/377588. PMC 1180678. PMID 12900798.
- ↑ Batini, Chiara; Hallast, Pille; Zadik, Daniel; Delser, Pierpaolo Maisano; Benazzo, Andrea; Ghirotto, Silvia; Arroyo-Pardo, Eduardo; Cavalleri, Gianpiero L.; De Knijff, Peter (2015). „Large-scale recent expansion of European patrilineages shown by population resequencing“. Nature Communications. 6: 7152. Bibcode:2015NatCo...6.7152B. doi:10.1038/ncomms8152. PMC 4441248. PMID 25988751.
- ↑ Mendez, L.; и др. (2016). „The Divergence of Neandertal and Modern Human Y Chromosomes“. The American Journal of Human Genetics. 98 (4): 728–34. doi:10.1016/j.ajhg.2016.02.023. PMC 4833433. PMID 27058445.
- ↑ „The father of all men is 340,000 years old“.
- ↑ „A recent bottleneck of Y chromosome diversity coincides with a global change in culture“. Genome Research. 25 (4): 459–466. April 2015. doi:10.1101/gr.186684.114. PMC 4381518. PMID 25770088.
- ↑ „A Rare Deep-Rooting D0 African Y-chromosomal Haplogroup and its Implications for the Expansion of Modern Humans Out of Africa“. Genetics. 212 (4): 1421–1428. June 2019. doi:10.1534/genetics.119.302368. PMC 6707464. PMID 31196864.
- ↑ Semino, Ornella; Magri, Chiara; Benuzzi, Giorgia; Lin, Alice A.; Al-Zahery, Nadia; Battaglia, Vincenza; MacCioni, Liliana; Triantaphyllidis, Costas; и др. (2004). „Origin, Diffusion, and Differentiation of Y-Chromosome Haplogroups E and J: Inferences on the Neolithization of Europe and Later Migratory Events in the Mediterranean Area“. The American Journal of Human Genetics. 74 (5): 1023–34. doi:10.1086/386295. PMC 1181965. PMID 15069642.
- ↑ 39,0 39,1 39,2 Semino, O; Magri, C; Benuzzi, G; и др. (May 2004). „Origin, diffusion, and differentiation of Y-chromosome haplogroups E and J: inferences on the neolithization of Europe and later migratory events in the Mediterranean area“. Am. J. Hum. Genet. 74 (5): 1023–34. doi:10.1086/386295. PMC 1181965. PMID 15069642.
- ↑ 40,0 40,1 40,2 40,3 40,4 40,5 40,6 Trombetta, Beniamino; d'Atanasio, Eugenia; Massaia, Andrea; Ippoliti, Marco; Coppa, Alfredo; Candilio, Francesca; Coia, Valentina; Russo, Gianluca; Dugoujon, Jean-Michel (2015). „Phylogeographic Refinement and Large Scale Genotyping of Human y Chromosome Haplogroup e Provide New Insights into the Dispersal of Early Pastoralists in the African Continent“. Genome Biology and Evolution. 7 (7): 1940–50. doi:10.1093/gbe/evv118. PMC 4524485. PMID 26108492.
- ↑ 41,0 41,1 Shi, Hong; Qi, Xuebin; Zhong, Hua; Peng, Yi; Zhang, Xiaoming; Ma, Runlin Z.; Su, Bing (2013). „Genetic Evidence of an East Asian Origin and Paleolithic Northward Migration of Y-chromosome Haplogroup N“. PLOS ONE. 8 (6): e66102. Bibcode:2013PLoSO...866102S. doi:10.1371/journal.pone.0066102. PMC 3688714. PMID 23840409.
- ↑ Batini, Chiara; Hallast, Pille; Zadik, Daniel; Delser, Pierpaolo Maisano; Benazzo, Andrea; Ghirotto, Silvia; Arroyo-Pardo, Eduardo; Cavalleri, Gianpiero L.; De Knijff, Peter (2015). „Large-scale recent expansion of European patrilineages shown by population resequencing“. Nature Communications. 6: 7152. Bibcode:2015NatCo...6.7152B. doi:10.1038/ncomms8152. PMC 4441248. PMID 25988751.
- ↑ 43,0 43,1 Rootsi, Siiri; и др. (2004). „Phylogeography of Y-Chromosome Haplogroup I Reveals Distinct Domains of Prehistoric Gene Flow in Europe“ (PDF). American Journal of Human Genetics. 75 (1): 128–37. doi:10.1086/422196. PMC 1181996. PMID 15162323. Архивирано од изворникот (PDF) на 2009-06-19. Посетено на 2016-05-04.
- ↑ Грешка во наводот: Погрешна ознака
<ref>; нема зададено текст за наводите по имеpau. - ↑ 45,0 45,1 Sharma, Swarkar; Rai, Ekta; Sharma, Prithviraj; Jena, Mamata; Singh, Shweta; Darvishi, Katayoon; Bhat, Audesh K.; Bhanwer, A J S.; Tiwari, Pramod Kumar (2009). „The Indian origin of paternal haplogroup R1a1* substantiates the autochthonous origin of Brahmins and the caste system“. Journal of Human Genetics. 54 (1): 47–55. doi:10.1038/jhg.2008.2. PMID 19158816.
- ↑ Jones, Eppie R.; Gonzalez-Fortes, Gloria; Connell, Sarah; Siska, Veronika; Eriksson, Anders; Martiniano, Rui; McLaughlin, Russell L.; Gallego Llorente, Marcos; Cassidy, Lara M. (2015). „Upper Palaeolithic genomes reveal deep roots of modern Eurasians“. Nature Communications. 6: 8912. Bibcode:2015NatCo...6.8912J. doi:10.1038/ncomms9912. PMC 4660371. PMID 26567969.
- ↑ 47,0 47,1 „Mesolithic Western Eurasian DNA“. Ancestral Journeys. Архивирано од изворникот на 2016-04-07. Посетено на 2016-05-04.
- ↑ 48,0 48,1 48,2 48,3 Грешка во наводот: Погрешна ознака
<ref>; нема зададено текст за наводите по имеcrucianii. - ↑ Evatt, Danny (2013-11-01). „The Evatt Clan: A Worldwide Historical Review of the Evatt Family Surname“.
- ↑ Sjödin, Per; François, Olivier (2011). „Wave-of-Advance Models of the Diffusion of the y Chromosome Haplogroup R1b1b2 in Europe“. PLOS ONE. 6 (6): e21592. Bibcode:2011PLoSO...621592S. doi:10.1371/journal.pone.0021592. PMC 3123369. PMID 21720564.
- ↑ 51,0 51,1 Underhill, Peter A.; Myres, Natalie M.; Rootsi, Siiri; Metspalu, Mait; Zhivotovsky, Lev A.; King, Roy J.; Lin, Alice A.; Chow, Cheryl-Emiliane T.; Semino, Ornella (2010). „Separating the post-Glacial coancestry of European and Asian y chromosomes within haplogroup R1a“. European Journal of Human Genetics. 18 (4): 479–84. doi:10.1038/ejhg.2009.194. PMC 2987245. PMID 19888303.CS1-одржување: display-автори (link)
- ^ 2005 Y-chromosome Phylogenetic Tree, from FamilyTreeДНК.com
- ^ A Nomenclature system for the Tree of Human Y-Chromosomal Haplogroups, Genome.org
Понатамошно читање[уреди | уреди извор]
- Mendez, Fernando; Krahn, Thomas; Schrack, Bonnie; Krahn, Astrid-Maria; Veeramah, Krishna; Woerner, August; Fomine, Forka Leypey Mathew; Bradman, Neil; Thomas, Mark (7 March 2013). „An African American paternal lineage adds an extremely ancient root to the human Y chromosome phylogenetic tree“ (PDF). American Journal of Human Genetics. 92 (3): 454–59. doi:10.1016/j.ajhg.2013.02.002. PMC 3591855. PMID 23453668. Архивирано од изворникот (PDF) на 2019-09-24. Посетено на 2024-02-09.
- „Y-Haplogroup A Phylogenetic Tree“. March 2013. Архивирано од изворникот на 2018-08-18. Посетено на 30 March 2013. (chart highlighting new branches added to the A phylotree in March 2013)
Надворешни врски[уреди | уреди извор]
- Дрво на хаплогрупата ISOGG Y-ДНК
- Семејно стебло ДНК Јавен хаплотре
- Табела на брзината на различни стапки на Y хромозомски STR мутации
- Карта на Y Хаплогрупи
- Атлас на човечкото патување, од Генографскиот проект, National Geographic
- Карта на Y-хаплогрупата на ДНК Херитиџ Архивирано на 18 февруари 2006 г.
- Видео туторијал за откривање на татковското потекло со Y-хромозоми
- Предвидувач на хаплогрупи
- Како PDF документ што ги дефинира хаплогрупите „Eu“.
- Y-ДНК хаплогрупи и проекти под-клада
- Описи, проекти и врски на Хаплоггрупата YДНК на Керхнер
- Компанија за тестирање на Y-ДНК Табела за споредба на маркери STR
- Y-ДНК Етнографски и генографски атлас и компилација на податоци со отворен код
- Y хромозомски конзорциум
