Античка ДНК

Античка ДНК (аДНК) — ДНК изолирана од антички примероци. [1] [2] Поради процесите на деградација (вклучувајќи вкрстено поврзување, деаминација и фрагментација) [3] античката ДНК е повеќе деградирана во споредба со современиот генетски материјал. [4] Дури и при најдобри услови за зачувување, постои горна граница од 0,4-1,5 милиони години за примерокот да содржи доволно ДНК за технологии за секвенционирање. [5] Најстариот примерок некогаш секвенциониран се проценува дека е стар 1,65 милиони години. [6] Генетскиот материјал е обновен од палео/археолошки и историски скелетни материјали, мумифицирани ткива, архивски збирки на незамрзнати медицински примероци, зачувани растителни остатоци, мраз и од морски и езерски седименти и нечистотија при ископување. На 7 декември 2022 година, Њујорк Тајмс објавил дека на Гренланд бил пронајден генетски материјал стар два милиони години и денеска се смета за најстарата ДНК откриена досега. [7] [8]
Историја на античките ДНК проучувања[уреди | уреди извор]
1980-ти[уреди | уреди извор]
Првото проучување за она кое подоцна било наречено аДНК било спроведено во 1984 година, кога Рас Хигучи и неговите колеги од Универзитетот во Калифорнија, Беркли објавиле дека трагите на ДНК од музејскиот примерок на квага не само што останале во примерокот повеќе од 150 години по смртта на поединецот, туку и дека може да се извлече и секвенционира. [9] Во текот на следните две години, преку истражувања на природни и вештачки мумифицирани примероци, Сванте Пабо потврдил дека овој феномен не е ограничен на релативно неодамнешните музејски примероци, туку очигледно може да се реплицира во низа мумифицирани човечки примероци кои датираат уште неколку илјади години. [10] [11] [12]
Макотрпните процеси кои биле потребни во тоа време за да се секвенционира таквата ДНК (преку бактериско клонирање) претставувале ефективна кочница за проучувањето на античката ДНК (аДНК) и полето на музеомијата. Меѓутоа, со развојот на полимеразната верижна реакција (PCR) во доцните 1980-ти, ова поле започнало брзо да напредува. [13] [14] [15] Двоен прајмер PCR засилување на аДНК (скокачко-PCR) може да произведе висока искривена и неавтентична секвенца на артефакти. За да се надминат тие недостатоци се користел повеќекратен прајмер, вгнездена PCR стратегија.
1990-ти[уреди | уреди извор]
Периодот на пос-ПЦР најавил бран на публикации бидејќи бројни истражувачки групи тврделе дека успеале да изолираат аДНК. Наскоро биле објавени низа неверојатни наоди, тврдејќи дека автентична ДНК може да се извлече од примероци стари милиони години, во доменот на она што Линдал (1993b) го означил како Антедилувска ДНК. [16] Поголемиот дел од таквите тврдења се засновале на пронаоѓање на ДНК од организми зачувани во килибар. Инсекти како што се пчели без осило, [17] [18] термити, [19] и дрвени комарци, [20] како и растителни [21] и бактериски [22] секвенци се вели дека биле извлечени од доминиканскиот килибар што датира од периодот на олигоцен. Сè уште постарите извори на либански килибари, кои датираат од периодот на креда, наводно, исто така дале автентична ДНК. [23] Тврдењата за пронаоѓање на ДНК не биле ограничени на килибар.
Биле објавени извештаи за неколку растителни остатоци зачувани со седимент, кои датираат од миоценот. [24] [25] Потоа во 1994 година, Вудворд и неговите соработници го објавиле она што во тоа време било наречено „највозбудливите резултати досега“ [26] - митохондриски цитохром б секвенци кои очигледно биле извлечени од коски од диносауруси кои датираат од пред повеќе од 80 милиони години. Кога во 1995 година, две дополнителни иследувања објавиле секвенци на ДНК на диносаурусите извлечени од јајце од периодот креда, [27] [28] се сметало дека полето ќе го револуционизира знаењето за еволутивното минато на Земјата. Дури и овие извонредни периоди биле на врвот со наводното пронаоѓање на 250 милиони години стари халобактериски секвенци од халит. [29] [30]
Развојот на подобро разбирање на кинетиката на зачувување на ДНК, ризиците од контаминација на примерокот и други комплицирачки фактори го навеле теренот да ги гледа овие резултати поскептично. Многубројните внимателни обиди не успеале да повторат многу од наодите и сите тврдења од деценијата за повеќемилионска аДНК биле отфрлени како неавтентични. [31]
2000-ти[уреди | уреди извор]
Во 2007 година било воведено засилување со единечен прајмер за да се реши оштетувањето на модификацијата на ДНК после смртта. [32] Од 2009 година полето на студиите за аДНК е револуционизирано со воведување на многу поевтини истражувачки техники. Употребата на техники за секвенционирање на следната генерација (NGS) со висока моќност во областа на истражување на древната ДНК е од суштинско значење за реконструкција на геномите на античките или изумрените организми. Методот на подготовка со едноверижна ДНК (ssДНК) предизвикал голем интерес кај античките истражувачи на ДНК (аДНК). [33] [34]
Покрај овие технички иновации, на почетокот на деценијата затеренот почнал да развива подобри стандарди и критериуми за евалуација на резултатите од ДНК, како и подобро разбирање на потенцијалните стапици. [35]
На 7 декември 2022 година, Њујорк Тајмс објавил дека на Гренланд бил пронајден генетски материјал стар два милиони години и денес се смета за најстарата ДНК откриена досега.
Проблеми и грешки[уреди | уреди извор]
Процеси на деградација[уреди | уреди извор]
Поради процесите на деградација (вклучувајќи вкрстено поврзување, деаминација и фрагментација), античката ДНК е со послаб квалитет од современиот генетски материјал. Карактеристиките на штетата и способноста на аДНК да преживее низ времето ги ограничува можните анализи и поставува горна граница на возраста на успешните примероци. Постои теоретска заемна поврзаност помеѓу времето и деградацијата на ДНК, [36] иако разликите во условите на животната средина ги комплицираат работите. Примероците подложени на различни услови веројатно нема предвидливо да се усогласат со со деградација на возраста. [37] Ефектите врз животната средина може дури и да бидат важни по ископувањето, бидејќи стапката на распаѓање на ДНК може да се зголеми, [38] особено при флуктуирачки услови на складирање. [39] Дури и при најдобри услови за зачувување, постои горна граница од 0,4 до 1,5 милиони години за примерокот да содржи доволно ДНК за современи технологии за секвенционирање.
Истражувањето за распаѓањето на митохондриската и нуклеарната ДНК во коските моа ја моделира деградацијата на митохондриската ДНК до просечна должина од 1 базен пар по 6.830.000 години на -5 °C. Кинетиката на распаѓање била измерена со експерименти со забрзано стареење, дополнително покажувајќи го силното влијание на температурата и влажноста на складирањето врз распаѓањето на ДНК. [40] Нуклеарната ДНК се разградува најмалку двапати побрзо од мтДНК. Раните студии кои објавиле обновување на многу постара ДНК, на пример од остатоци од диносаурус од креда, можеби произлегле од контаминација на примерокот.
Возрасна граница[уреди | уреди извор]
Критичкиот преглед на античката ДНК литература преку развојот на ова поле, нагласува дека неколку проучувања успеале да ја засилат ДНК од остатоците постари од неколку стотици илјади години. [41] Поголемата благодарност за ризиците од контаминација на животната средина и иследувањата за хемиската стабилност на ДНК предизвикале загриженост во однос на претходно пријавените резултати. Наводната ДНК на диносаурусот подоцна било откриено дека е човечки Y-хромозом. [42] ДНК пријавена од инкапсулирани халобактерии била критикувана врз основа на нејзината сличност со современите бактерии, што укажува на контаминација, или тие може да се производ на долготрајна метаболичка активност на ниско ниво. [43]
аДНК би можела да содржи поголем број на постмортални мутации, кои се зголемуваат со текот на времето. Некои региони на полинуклеотид се поподложни на оваа деградација, дозволувајќи им на податоците од погрешната низа да ги заобиколат статистичките филтри што се користат за проверка на валидноста на податоците. Поради честите грешки во секвенционирањето, потребно е да се примени голема претпазливост при толкувањето на големината на популацијата. [44] Замените кои произлегуваат од деаминацијата на остатоците од цитозин се многу презастапени во античките секвенци на ДНК. Погрешното кодирање на C во T и G до A е одговорно за повеќето грешки. [45]
Контаминација[уреди | уреди извор]
Друг проблем со древните примероци на ДНК е контаминација со современата човечка ДНК и со микробна ДНК (од кои повеќето се исто така антички). [46] [47] Во последниве години се појавиле нови методи за да се спречи можна контаминација на примероците од аДНК, вклучително спроведување на екстракција во екстремни стерилни услови, користење специјални апарати за идентификување на ендогени молекули на примерокот (се разликуваат од оние воведени за време на анализата) и примена на биоинформатика на добиените секвенци врз основа на познати читања со цел да се приближат стапките на контаминација. [48] [49]
Автентикација на аДНК[уреди | уреди извор]
Развојот на полето на аДНК во 2000-тите ја зголемило важноста на оригиналноста на обновената ДНК за да се потврди дека таа е навистина стара и не претставува резултат на неодамнешна контаминација. Како што ДНК се разградува со текот на времето, нуклеотидите кои ја сочинуваат ДНК може да се променат, особено на краевите на молекулите на ДНК. Деаминацијата на цитозин во урацил на краевите на молекулите на ДНК станало начин за автентикација. За време на секвенционирањето на ДНК, ДНК полимеразите ќе инкорпорираат аденин (А) наспроти урацилот (U), што доведува до замена на цитозин (C) во тимин (Т) во податоците на аДНК. [50] Овие замени се зголемуваат во фреквенцијата како што примерокот старее. Мерењето на фреквенцијата на нивото на КТ, древно оштетување на ДНК, може да се спроведе со користење на различен софтвер како mapDamage2.0 или PMDtools [51] [52] и интерактивно на metaDMG. [53] Поради хидролитичка депуринација, ДНК фрагменти на помали парчиња доведува до едноверижни паузи. Во комбинација со моделот на оштетување, оваа кратка должина на фрагментот исто така може да помогне да се направи разлика помеѓу современата и античката ДНК. [54] [55]
Нечовечка аДНК[уреди | уреди извор]
И покрај проблемите поврзани со „преддилувијанската“ ДНК, денес се објавени широк и сè поголем опсег на секвенци на аДНК од низа животински и растителни таксони. Испитувачките ткива вклучуваат вештачки или природно мумифицирани животински остатоци, [56] коски, [57] [58] [59] [60] палеофецеси, [61] [62] примероци зачувани со алкохол, [63] средини од глодар, [64] сушени растителни остатоци, [65] [66] и неодамна, екстракција на животинска и растителна ДНК директно од примероците од почвата. [67]
Во јуни 2013 година, група истражувачи, вклучувајќи ги Еске Вилерслев, Маркус Томас Гилберт и Орландо Лудович од Центарот за геогенетика на Природонаучниот музеј на Данска одУниверзитетот во Копенхаген, објавиле дека ја секвенционирале ДНК од коњ кој бил стар помеѓу 560-780 илјади години, користејќи материјал извлечен од коска од ногата пронајдена закопана во вечен мраз на канадската територија Јукон. [68] [69] [70] Германски тим, исто така, во 2013 година пријавил реконструиран митохондриски геном на мечка, Ursus deningeri, стар повеќе од 300.000 години, докажувајќи дека автентичната античка ДНК може да се зачува стотици илјади години надвор од вечниот мраз. [71] ДНК секвенцата на уште постара нуклеарна ДНК била пријавена во 2021 година од зачуваните во вечниот мраз заби на два сибирски мамути, и двата стари над милион години. [72]
Истражувачите во 2016 година измериле ДНК на хлоропласт во јадрата на морските седименти и откриле ДНК на силикатна алга која датира од 1,4 милиони години. [73] Оваа ДНК имала полуживот значително подолг од претходните истражувања, до 15.000 години. Екипата на Киркпатрик, исто така, открила дека ДНК се распаѓа само со стапка на полуживот до околу 100 илјади години, во тој момент таа следела побавна стапка на распаѓање со законска моќност.
Човечка аДНК[уреди | уреди извор]
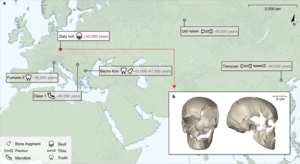
Поради значителниот антрополошки, археолошки и јавен интерес насочен кон човечките остатоци, тие добиле значително внимание од заедницата на ДНК. Постојат и подлабоки прашања со контаминација, бидејќи примероците припаѓаат на истиот вид како и истражувачите кои ги собираат и оценуваат примероците.
Извори[уреди | уреди извор]
Поради морфолошката зачувување во мумиите, многу проучувања од 1990-тите и 2000-тите користеле мумифицирано ткиво како извор на античка човечка ДНК. Примерите вклучуваат и природно сочувани примероци, како што е ледениот човек Еци замрзнат во глечер [74] и тела зачувани преку брзо исушување на голема надморска височина на Андите, [75] како и разни хемиски обработени зачувани ткива како што се мумии на Стар Египет. [76] Сепак, мумифицираните остатоци се со ограничен ресурс. Поголемиот дел од иследувањата на човечката аДНК се фокусирале на екстракција на ДНК од два извори многу почести во археолошките записи: коски и заби. Коската која најчесто се користи за екстракција на ДНК е петролестата ушна коска, бидејќи нејзината густа структура обезбедува добри услови за зачувување на ДНК. [77] Неколку други извори, исто така, дале ДНК, вклучувајќи палеофецеси, [78] и влакна. [79] Контаминацијата останува главен проблем кога се работи на антички човечки материјал.
ДНК на антички патоген била успешно извлечена од примероци стари повеќе од 5.000 години кај луѓето и пред 17.000 години кај други видови. Покрај вообичаените извори на мумифицирано ткиво, коски и заби, ваквите иследувања испитувале и низа други примероци од ткиво, вклучувајќи калцифицирана плевра, [80] ткиво вградено во парафин, [81] [82] и ткиво фиксирано со формалин. [83] Биле развиени ефикасни компјутерски алатки за аДНК анализи на патогени и микроорганизми во мал (QIIME [84]) и голем размер (FALCON [85]).
Резултати[уреди | уреди извор]
Сепак, преземајќи превентивни мерки во нивната постапка против таквата контаминација, проучување од 2012 година анализирало примероци од коски од неандерталска група во пештерата ел-Сидрон, наоѓајќи нови сознанија за потенцијалното сродство и генетската разновидност од аДНК. [86] Во ноември 2015 година, научниците објавиле дека пронашле заб стар 110.000 години кој содржи ДНК од Денисовскиот хоминин, изумрен вид на човек од родот Хомо. [87] [88]
Истражувањето додало нова сложеност на жителите на Евроазија. Иследување од 2018 година [89] покажало дека масовна миграција во бронзеното време во голема мера влијаела на генетскиот состав на Британските Острови, носејќи ја со себе културата на ѕвонестите пехари од континентална Европа.
Исто така, истражувањето открило нови информации за врските меѓу предците на Средна Азија и домородните народи на Америка. Во Африка, постарата ДНК брзо се разградува поради потоплата тропска клима, иако во септември 2017 година биле пријавени древни примероци на ДНК, стари дури 8.100 години. [90]
Покрај тоа, древната ДНК им помогнала на истражувачите да ја проценат современата човечка дивергенција. [91] Со секвенционирање на африканските геноми од тројца ловци-собирачи од камено доба (стари 2000 години) и четворица земјоделци од железното време (стари 300 до 500 години), Шлебуш и неговите колеги успеале да го вратат датумот на најраната дивергенција меѓу човечката популација на 350.000 до 260.000 години.
Од 2021 година, најстарите целосно реконструирани човечки геноми се стари околу 45.000 години.[92] Ваквите генетски податоци обезбедуваат увид во миграцијата и генетската историја - на пр. на Европа - вклучително и за вкрстувањето помеѓу архаичните и модерните луѓе како вообичаена мешавина помеѓу почетните европски современи луѓе и неандерталците. [93]
Наводи[уреди | уреди извор]
- ↑ Pevsner J (2015). Bioinformatics and Functional Genomics (3rd. изд.). Wiley-Blackwell. ISBN 978-1118581780.
- ↑ Jones M (2016). Unlocking the Past: How Archaeologists Are Rewriting Human History with Ancient DNA. Arcade. ISBN 978-1628724479.
- ↑ „A chemical framework for the preservation of fossil vertebrate cells and soft tissues“. Earth-Science Reviews. 240: 104367. May 2023. doi:10.1016/j.earscirev.2023.104367.
- ↑ „The half-life of DNA in bone: measuring decay kinetics in 158 dated fossils“. Proceedings. Biological Sciences. 279 (1748): 4724–33. December 2012. doi:10.1098/rspb.2012.1745. PMC 3497090. PMID 23055061.CS1-одржување: display-автори (link)
- ↑ „Long-term persistence of bacterial DNA“ (PDF). Current Biology. 14 (1): R9-10. January 2004. doi:10.1016/j.cub.2003.12.012. PMID 14711425.CS1-одржување: display-автори (link)
- ↑ „Million-year-old DNA sheds light on the genomic history of mammoths“. Nature. 591 (7849): 265–269. March 2021. Bibcode:2021Natur.591..265V. doi:10.1038/s41586-021-03224-9. PMC 7116897. PMID 33597750 Проверете ја вредноста
|pmid=(help).CS1-одржување: display-автори (link) - ↑ Zimmer, Carl (7 December 2022). „Oldest Known DNA Offers Glimpse of a Once-Lush Arctic - In Greenland's permafrost, scientists discovered two-million-year-old genetic material from scores of plant and animal species, including mastodons, geese, lemmings and ants“. The New York Times. Посетено на 7 December 2022.
- ↑ Kjær, Kurt H.; и др. (7 December 2022). „A 2-million-year-old ecosystem in Greenland uncovered by environmental DNA“. Nature. 612 (7939): 283–291. Bibcode:2022Natur.612..283K. doi:10.1038/s41586-022-05453-y. PMC 9729109 Проверете ја вредноста
|pmc=(help). PMID 36477129 Проверете ја вредноста|pmid=(help). - ↑ „DNA sequences from the quagga, an extinct member of the horse family“. Nature. 312 (5991): 282–4. 1984. Bibcode:1984Natur.312..282H. doi:10.1038/312282a0. PMID 6504142.
- ↑ „Preservation of DNA in ancient Egyptian mummies“. J. Archaeol. Sci. 12 (6): 411–17. 1985a. doi:10.1016/0305-4403(85)90002-0.
- ↑ „Molecular cloning of Ancient Egyptian mummy DNA“. Nature. 314 (6012): 644–5. 1985b. Bibcode:1985Natur.314..644P. doi:10.1038/314644a0. PMID 3990798.
- ↑ „Molecular genetic investigations of ancient human remains“. Cold Spring Harbor Symposia on Quantitative Biology. 51 (Pt 1): 441–6. 1986. doi:10.1101/SQB.1986.051.01.053. PMID 3107879.
- ↑ Mullis KB, Faloona FA (1987). Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction. Methods in Enzymology. 155. стр. 335–50. doi:10.1016/0076-6879(87)55023-6. ISBN 978-0-12-182056-5. PMID 3431465.
- ↑ Raxworthy, Christopher J.; Smith, Brian Tilston (November 2021). „Mining museums for historical DNA: advances and challenges in museomics“ (PDF). Trends in Ecology & Evolution. 36 (11): 1049–1060. doi:10.1016/j.tree.2021.07.009. PMID 34456066 Проверете ја вредноста
|pmid=(help). Посетено на 27 June 2022. - ↑ „Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase“. Science. 239 (4839): 487–91. January 1988. Bibcode:1988Sci...239..487S. doi:10.1126/science.239.4839.487. PMID 2448875.CS1-одржување: display-автори (link)
- ↑ „Recovery of antediluvian DNA“. Nature. 365 (6448): 700. October 1993. Bibcode:1993Natur.365..700L. doi:10.1038/365700a0. PMID 8413647.
- ↑ „Isolation and partial characterisation of DNA from the bee Problebeia dominicana (Apidae:Hymenoptera) in 25–40 million year old amber“. Med Sci Res. 20: 249–51. 1992a.
- ↑ „Enzymatic amplification and nucleotide sequencing of portions of the 18S rRNA gene of the bee Problebeia dominicana (Apidae:Hymenoptera) isolated from 25–40 million year old Dominican amber“. Med Sci Res. 20: 619–22. 1992b.
- ↑ „Extracting DNA from the gut microbes of the termite (Zootermopsis nevadensis)“. Journal of Visualized Experiments (4): 195. 2007. doi:10.3791/195. PMC 2556161. PMID 18979000.
- ↑ „Very old DNA“. Current Opinion in Genetics & Development. 4 (6): 810–5. December 1994. doi:10.1016/0959-437x(94)90064-7. PMID 7888749.
- ↑ „DNA from an extinct plant“. Nature. 363 (6431): 677. 1993. Bibcode:1993Natur.363..677P. doi:10.1038/363677a0.
- ↑ „Bacillus DNA in fossil bees: an ancient symbiosis?“. Applied and Environmental Microbiology. 60 (6): 2164–2167. June 1994. Bibcode:1994ApEnM..60.2164C. doi:10.1128/aem.60.6.2164-2167.1994. PMC 201618. PMID 8031102.
- ↑ „Amplification and sequencing of DNA from a 120-135-million-year-old weevil“. Nature. 363 (6429): 536–538. June 1993. Bibcode:1993Natur.363..536C. doi:10.1038/363536a0. PMID 8505978.
- ↑ „Amplification and analysis of Miocene plant fossil DNA“. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 333 (1268): 419–26, discussion 426–7. September 1991. doi:10.1098/rstb.1991.0092. PMID 1684052.
- ↑ „Chloroplast DNA sequence from a miocene Magnolia species“. Nature. 344 (6267): 656–8. April 1990. Bibcode:1990Natur.344..656G. doi:10.1038/344656a0. PMID 2325772.
- ↑ „DNA sequence from Cretaceous period bone fragments“. Science. 266 (5188): 1229–32. November 1994. Bibcode:1994Sci...266.1229W. doi:10.1126/science.7973705. PMID 7973705.
- ↑ „Molecular cloning and sequencing of the 18S rDNA from specialized dinosaur egg fossil found in Xixia Henan, China“. Acta Sci Nat Univ Pekinensis. 31: 140–47. 1995.
- ↑ „DNA isolation and sequence analysis of dinosaur DNA from Cretaceous dinosaur egg in Xixia Henan, China“. Acta Sci Nat Univ Pekinensis. 31: 148–52. 1995.
- ↑ „Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal“. Nature. 407 (6806): 897–900. October 2000. Bibcode:2000Natur.407..897V. doi:10.1038/35038060. PMID 11057666.
- ↑ „Recovery of 16S ribosomal RNA gene fragments from ancient halite“. Nature. 417 (6887): 432–6. May 2002. Bibcode:2002Natur.417..432F. doi:10.1038/417432a. PMID 12024211.
- ↑ „Genetic analyses from ancient DNA“ (PDF). Annual Review of Genetics. 38 (1): 645–79. 2004. doi:10.1146/annurev.genet.37.110801.143214. PMID 15568989. Архивирано од изворникот (PDF) на December 17, 2008.CS1-одржување: display-автори (link)
- ↑ „Novel high-resolution characterization of ancient DNA reveals C > U-type base modification events as the sole cause of post mortem miscoding lesions“. Nucleic Acids Research. 35 (17): 5717–28. 2007. doi:10.1093/nar/gkm588. PMC 2034480. PMID 17715147.
- ↑ „New insights on single-stranded versus double-stranded DNA library preparation for ancient DNA“. BioTechniques. 59 (6): 368–71. December 2015. doi:10.2144/000114364. PMID 26651516.CS1-одржување: display-автори (link)
- ↑ „Library construction for ancient genomics: single strand or double strand?“. BioTechniques. 56 (6): 289–90, 292–6, 298, passim. June 2014. doi:10.2144/000114176. PMID 24924389.
- ↑ „Ancient DNA comes of age“. PLOS Biology. 3 (2): e56. February 2005. doi:10.1371/journal.pbio.0030056. PMC 548952. PMID 15719062.
- ↑ „Geologically ancient DNA: fact or artefact?“. Trends in Microbiology. 13 (5): 212–20. May 2005. doi:10.1016/j.tim.2005.03.010. PMID 15866038.
- ↑ „Crosslinks rather than strand breaks determine access to ancient DNA sequences from frozen sediments“. Genetics. 173 (2): 1175–9. June 2006. doi:10.1534/genetics.106.057349. PMC 1526502. PMID 16582426.CS1-одржување: display-автори (link)
- ↑ „Freshly excavated fossil bones are best for amplification of ancient DNA“. Proceedings of the National Academy of Sciences of the United States of America. 104 (3): 739–44. January 2007. Bibcode:2007PNAS..104..739P. doi:10.1073/pnas.0610257104. PMC 1783384. PMID 17210911.CS1-одржување: display-автори (link)
- ↑ „DNA preservation: a microsatellite-DNA study on ancient skeletal remains“. Electrophoresis. 20 (8): 1722–8. June 1999. doi:10.1002/(sici)1522-2683(19990101)20:8<1722::aid-elps1722>3.0.co;2-4. PMID 10435438.
- ↑ „Robust chemical preservation of digital information on DNA in silica with error-correcting codes“. Angewandte Chemie. 54 (8): 2552–5. February 2015. doi:10.1002/anie.201411378. PMID 25650567.
- ↑ „Diverse plant and animal genetic records from Holocene and Pleistocene sediments“. Science. 300 (5620): 791–5. May 2003. Bibcode:2003Sci...300..791W. doi:10.1126/science.1084114. PMID 12702808.CS1-одржување: display-автори (link)
- ↑ „Detecting dinosaur DNA“. Science. 268 (5214): 1192–3, author reply 1194. May 1995. Bibcode:1995Sci...268.1191B. doi:10.1126/science.7605504. PMID 7605504.
- ↑ „Ancient bacteria show evidence of DNA repair“. Proceedings of the National Academy of Sciences of the United States of America. 104 (36): 14401–5. September 2007. Bibcode:2007PNAS..10414401J. doi:10.1073/pnas.0706787104. PMC 1958816. PMID 17728401.CS1-одржување: display-автори (link)
- ↑ „Accounting for bias from sequencing error in population genetic estimates“. Molecular Biology and Evolution (Free full text)
|format=бара|url=(help). 25 (1): 199–206. January 2008. doi:10.1093/molbev/msm239. PMID 17981928. - ↑ „Patterns of damage in genomic DNA sequences from a Neandertal“. Proceedings of the National Academy of Sciences of the United States of America. 104 (37): 14616–21. September 2007. Bibcode:2007PNAS..10414616B. doi:10.1073/pnas.0704665104. PMC 1976210. PMID 17715061.CS1-одржување: display-автори (link)
- ↑ „Selective enrichment of damaged DNA molecules for ancient genome sequencing“. Genome Research. 24 (9): 1543–9. September 2014. doi:10.1101/gr.174201.114. PMC 4158764. PMID 25081630.
- ↑ „Metagenomic Composition Analysis of an Ancient Sequenced Polar Bear Jawbone from Svalbard“. Genes. 9 (9): 445. September 2018. doi:10.3390/genes9090445. PMC 6162538. PMID 30200636.CS1-одржување: display-автори (link)
- ↑ „Ancient DNA and human history“. Proceedings of the National Academy of Sciences of the United States of America. 113 (23): 6380–7. June 2016. Bibcode:2016PNAS..113.6380S. doi:10.1073/pnas.1524306113. PMC 4988579. PMID 27274045.
- ↑ „PyDamage: automated ancient damage identification and estimation for contigs in ancient DNA de novo assembly“. PeerJ. 9: e11845. 2021-07-27. doi:10.7717/peerj.11845. PMC 8323603 Проверете ја вредноста
|pmc=(help). PMID 34395085 Проверете ја вредноста|pmid=(help). - ↑ Dabney, Jesse; Meyer, Matthias; Pääbo, Svante (1 July 2013). „Ancient DNA Damage“. Cold Spring Harbor Perspectives in Biology. 5 (7): a012567. doi:10.1101/cshperspect.a012567. PMC 3685887. PMID 23729639.
- ↑ Jónsson, H; Ginolhac, A; Schubert, M; Johnson, PL; Orlando, L (1 July 2013). „mapDamage2.0: fast approximate Bayesian estimates of ancient DNA damage parameters“. Bioinformatics. 29 (13): 1682–4. doi:10.1093/bioinformatics/btt193. PMC 3694634. PMID 23613487.
- ↑ Skoglund, P; Northoff, BH; Shunkov, MV; Derevianko, AP; Pääbo, S; Krause, J; Jakobsson, M (11 February 2014). „Separating endogenous ancient DNA from modern day contamination in a Siberian Neandertal“. Proceedings of the National Academy of Sciences of the United States of America. 111 (6): 2229–34. doi:10.1073/pnas.1318934111. PMC 3926038. PMID 24469802.
- ↑ Michelsen, Christian; Pedersen, Mikkel Winther; Fernandez-Guerra, Antonio; Zhao, Lei; Petersen, Troels C.; Korneliussen, Thorfinn Sand (9 December 2022). „metaDMG – A Fast and Accurate Ancient DNA Damage Toolkit for Metagenomic Data“: 2022.12.06.519264. doi:10.1101/2022.12.06.519264. Наводот journal бара
|journal=(help) - ↑ Krause, Johannes; Briggs, Adrian W.; Kircher, Martin; Maricic, Tomislav; Zwyns, Nicolas; Derevianko, Anatoli; Pääbo, Svante (9 February 2010). „A Complete mtDNA Genome of an Early Modern Human from Kostenki, Russia“. Current Biology. 20 (3): 231–236. doi:10.1016/j.cub.2009.11.068. PMID 20045327.
- ↑ Pochon, Zoé; Bergfeldt, Nora; Kırdök, Emrah; Vicente, Mário; Naidoo, Thijessen; Valk, Tom van der; Altınışık, N. Ezgi; Krzewińska, Maja; Dalen, Love (5 October 2022). „aMeta: an accurate and memory-efficient ancient Metagenomic profiling workflow“: 2022.10.03.510579. doi:10.1101/2022.10.03.510579. Наводот journal бара
|journal=(help) - ↑ „DNA phylogeny of the extinct marsupial wolf“. Nature. 340 (6233): 465–7. August 1989. Bibcode:1989Natur.340..465T. doi:10.1038/340465a0. PMID 2755507.
- ↑ „Ancient bone DNA amplified“. Nature. 342 (6249): 485. November 1989. Bibcode:1989Natur.342..485H. doi:10.1038/342485a0. PMID 2586623.
- ↑ „Independent origins of New Zealand moas and kiwis“. Proceedings of the National Academy of Sciences of the United States of America. 89 (18): 8741–4. September 1992. Bibcode:1992PNAS...89.8741C. doi:10.1073/pnas.89.18.8741. PMC 49996. PMID 1528888.
- ↑ „DNA from ancient mammoth bones“. Nature. 370 (6488): 333–4. August 1994. Bibcode:1994Natur.370R.333H. doi:10.1038/370333b0. PMID 8047136.
- ↑ „Tracking the origins of the cave bear (Ursus spelaeus) by mitochondrial DNA sequencing“. Proceedings of the National Academy of Sciences of the United States of America. 91 (25): 12336–40. December 1994. Bibcode:1994PNAS...9112336H. doi:10.1073/pnas.91.25.12336. PMC 45432. PMID 7991628.
- ↑ „Molecular coproscopy: dung and diet of the extinct ground sloth Nothrotheriops shastensis“. Science. 281 (5375): 402–6. July 1998. Bibcode:1998Sci...281..402P. doi:10.1126/science.281.5375.402. PMID 9665881.CS1-одржување: display-автори (link)
- ↑ „A molecular analysis of ground sloth diet through the last glaciation“. Molecular Ecology. 9 (12): 1975–84. December 2000. doi:10.1046/j.1365-294X.2000.01106.x. PMID 11123610.
- ↑ „Methods for the recovery of mitochondrial DNA sequences from museum specimens of myiasis-causing flies“. Medical and Veterinary Entomology. 16 (1): 39–45. March 2002. doi:10.1046/j.0269-283x.2002.00336.x. PMID 11963980.
- ↑ „Molecular analysis of an 11,700-year-old rodent midden from the Atacama Desert, Chile“. Molecular Ecology. 11 (5): 913–24. May 2002. doi:10.1046/j.1365-294X.2002.01492.x. PMID 11975707.
- ↑ „Evolution of maize inferred from sequence diversity of an Adh2 gene segment from archaeological specimens“. Proceedings of the National Academy of Sciences of the United States of America. 90 (5): 1997–2001. March 1993. Bibcode:1993PNAS...90.1997G. doi:10.1073/pnas.90.5.1997. PMC 46007. PMID 8446621.
- ↑ „Amplification of oak DNA from ancient and modern wood“. Molecular Ecology. 8 (12): 2137–40. December 1999. doi:10.1046/j.1365-294x.1999.00788.x. PMID 10632865.
- ↑ „Ancient DNA“. Proceedings. Biological Sciences. 272 (1558): 3–16. January 2005. doi:10.1098/rspb.2004.2813. PMC 1634942. PMID 15875564.
- ↑ Erika Check Hayden (26 June 2013). „First horses arose 4 million years ago“. Nature. doi:10.1038/nature.2013.13261.
- ↑ „World's Oldest Genome Sequenced From 700,000-Year-Old Horse DNA“. National Geographic. November 7, 2017. Посетено на May 19, 2019.
- ↑ „Recalibrating Equus evolution using the genome sequence of an early Middle Pleistocene horse“. Nature. 499 (7456): 74–8. July 2013. Bibcode:2013Natur.499...74O. doi:10.1038/nature12323. PMID 23803765.CS1-одржување: display-автори (link)
- ↑ „Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments“. Proceedings of the National Academy of Sciences of the United States of America. 110 (39): 15758–63. September 2013. Bibcode:2013PNAS..11015758D. doi:10.1073/pnas.1314445110. PMC 3785785. PMID 24019490.CS1-одржување: display-автори (link)
- ↑ „Million-year-old mammoth genomes shatter record for oldest ancient DNA“. Nature. 590 (7847): 537–538. February 2021. Bibcode:2021Natur.590..537C. doi:10.1038/d41586-021-00436-x. ISSN 0028-0836. PMID 33597786 Проверете ја вредноста
|pmid=(help). - ↑ „Fossil DNA persistence and decay in marine sediment over hundred-thousand-year to million-year time scales“. Geology (англиски). 44 (8): 615–18. 2016-07-08. Bibcode:2016Geo....44..615K. doi:10.1130/g37933.1. ISSN 0091-7613.
- ↑ „Molecular genetic analyses of the Tyrolean Ice Man“. Science. 264 (5166): 1775–8. June 1994. Bibcode:1994Sci...264.1775H. doi:10.1126/science.8209259. PMID 8209259.CS1-одржување: display-автори (link)
- ↑ „Authenticating ancient human mitochondrial DNA“. Human Biology. 73 (5): 689–713. October 2001. doi:10.1353/hub.2001.0069. PMID 11758690.
- ↑ „An unusual mitochondrial DNA sequence variant from an Egyptian mummy“. Genomics. 22 (2): 487–9. July 1994. doi:10.1006/geno.1994.1417. PMID 7806242.
- ↑ „Optimal Ancient DNA Yields from the Inner Ear Part of the Human Petrous Bone“. PLOS ONE. 10 (6): e0129102. 2015-06-18. Bibcode:2015PLoSO..1029102P. doi:10.1371/journal.pone.0129102. PMC 4472748. PMID 26086078.CS1-одржување: display-автори (link)
- ↑ „A molecular analysis of dietary diversity for three archaic Native Americans“. Proceedings of the National Academy of Sciences of the United States of America. 98 (8): 4317–22. April 2001. Bibcode:2001PNAS...98.4317P. doi:10.1073/pnas.061014798. PMC 31832. PMID 11296282.CS1-одржување: display-автори (link)
- ↑ „Ancient mitochondrial DNA from hair“. Current Biology. 14 (12): R463-4. June 2004. doi:10.1016/j.cub.2004.06.008. PMID 15203015.CS1-одржување: display-автори (link)
- ↑ „Mycobacterium tuberculosis complex DNA in calcified pleura from remains 1400 years old“. Lett Appl Microbiol. 27 (5): 265–69. 1998. doi:10.1046/j.1472-765x.1998.t01-8-00449.x. PMID 9830142.
- ↑ „PCR analysis of tissue samples from the 1979 Sverdlovsk anthrax victims: the presence of multiple Bacillus anthracis strains in different victims“. Proceedings of the National Academy of Sciences of the United States of America. 95 (3): 1224–9. February 1998. Bibcode:1998PNAS...95.1224J. doi:10.1073/pnas.95.3.1224. PMC 18726. PMID 9448313.CS1-одржување: display-автори (link)
- ↑ „Sequence of the 1918 pandemic influenza virus nonstructural gene (NS) segment and characterization of recombinant viruses bearing the 1918 NS genes“. Proceedings of the National Academy of Sciences of the United States of America. 98 (5): 2746–51. February 2001. Bibcode:2001PNAS...98.2746B. doi:10.1073/pnas.031575198. PMC 30210. PMID 11226311.CS1-одржување: display-автори (link)
- ↑ „Initial genetic characterization of the 1918 "Spanish" influenza virus“. Science. 275 (5307): 1793–6. March 1997. doi:10.1126/science.275.5307.1793. PMID 9065404.
- ↑ QIIME
- ↑ „FALCON: a method to infer metagenomic composition of ancient DNA“. bioRxiv. February 2018. doi:10.1101/267179.
- ↑ „Palaeogenetic research at the El Sidrón Neanderthal site“. Annals of Anatomy - Anatomischer Anzeiger. Special Issue: Ancient DNA. 194 (1): 133–7. January 2012. doi:10.1016/j.aanat.2011.01.014. PMID 21482084.
- ↑ „In a Tooth, DNA From Some Very Old Cousins, the Denisovans“. The New York Times. 16 November 2015. Посетено на 16 November 2015.
- ↑ „Nuclear and mitochondrial DNA sequences from two Denisovan individuals“. Proceedings of the National Academy of Sciences of the United States of America. 112 (51): 15696–700. December 2015. Bibcode:2015PNAS..11215696S. doi:10.1073/pnas.1519905112. PMC 4697428. PMID 26630009.CS1-одржување: display-автори (link)
- ↑ „The Beaker phenomenon and the genomic transformation of northwest Europe“. Nature. 555 (7695): 190–196. March 2018. Bibcode:2018Natur.555..190O. doi:10.1038/nature25738. PMC 5973796. PMID 29466337.CS1-одржување: display-автори (link)
- ↑ „Clues to Africa's Mysterious Past Found in Ancient Skeletons“. The New York Times. 21 September 2017. Посетено на 21 September 2017.
- ↑ „Southern African ancient genomes estimate modern human divergence to 350,000 to 260,000 years ago“. Science. 358 (6363): 652–655. November 2017. Bibcode:2017Sci...358..652S. doi:10.1126/science.aao6266. PMID 28971970.CS1-одржување: display-автори (link)
- ↑ „Neanderthal ancestry identifies oldest modern human genome“. phys.org (англиски). Посетено на 10 May 2021.
- ↑ „Europe's oldest known humans mated with Neandertals surprisingly often“. Science News. 7 April 2021. Посетено на 10 May 2021.
Надворешни врски[уреди | уреди извор]
- „Ancient DNA“. Ancestral journeys. Архивирано од изворникот на October 3, 2016.
- Famous mtDNA, isogg wiki
- Ancient mtDNA, isogg wiki
- Ancient DNA Архивирано на {{{2}}}., y-str.org
- Evidence of the Past: A Map and Status of Ancient Remains – samples from USA no sequence data here.
- „Unravelling the mummy mystery – using DNA“. Архивирано од изворникот на December 14, 2009 – no data on YDNA only mtDNA
